Revisión
Asociación entre COVID-19 e infecciones bacterianas. Una revisión de la literatura
Association between COVID-19 and bacterial infection. A literature review
Actual Med.2025;110(821):9-21 DOI: 10.15568/am.2025.821.rev01
Recibido: 31/07/2024
Revisado: 15/09/2024
Aceptado: 18/12/2024
Resumen
El SARS-CoV-2 es el agente causal de la enfermedad COVID-19, que ha impactado todos los ámbitos de la sociedad, en especial en el periodo pandémico entre 2020 – 2023. La presentación clínica de COVID-19 es variable, con casos asintomático o de resolución favorable. Una complicación de los pacientes graves por COVID-19 es la infección concomitante con otros patógenos, destacando las infecciones bacterianas con cepas multirresistentes. En este trabajo se realizó una revisión para investigar el impacto de la pandemia en los sistemas de salud en cuanto a la concurrencia de infecciones SARS-CoV-2 y otros patógenos bacterianos, así como en el problema de resistencia a los antimicrobianos.
Los resultados de esta revisión evidencian que las coinfecciones se presentan principalmente en pacientes con comorbilidades y con un cuadro más grave de COVID-19. Esto implica una mayor necesidad de cuidados especiales y larga estancia hospitalaria, con aumento de la posibilidad de complicaciones e infecciones asociadas al cuidado de la salud. Además, se detectó un uso indiscriminado de antibióticos por parte del personal clínico como tratamiento inicial y, debido la excesiva atención requerida por la pandemia, se descuidaron los programas de vigilancia epidemiológica a la resistencia antimicrobiana.
En los perfiles de resistencia antimicrobiana, se evidenció la presencia de mecanismos de resistencia que incluyen el aumento en el reporte de MRSA, bacilos Gram negativos con BLEE o con NDM, con alta resistencia a carbapenémicos, a la polimixina B, entre otros. De los organismos, sobresalen Acinetobacter. baumannii, Escherichia coli, Pseudomonas aeruginosa, Klebsiella pneumoniae, especies de los géneros Enterococcus y Streptococcus, Staphylococcus aureus incluyendo MRSA, entre otros.
En conjunto, se evidencia la necesidad de seguir monitoreando, evaluar los aislamientos y establecer los protocolos necesarios para brindar un manejo adecuado de la concurrencia de infecciones y monitoreo de la resistencia a los antimicrobianos a nivel local y regional, en especial ante eventos epidemiológicos de gran magnitud como ha sido la pandemia por COVID-19.
Palabras clave: COVID-19; Coinfecciones; Bacterias; SARS-CoV-2; Resistencia antimicrobiana.
ABSTRACT
SARS-CoV-2 is the causative agent of COVID-19, which has impacted all aspects of society, especially during the pandemic period between 2020 – 2023. The clinical presentation of COVID-19 is variable, with asymptomatic cases or favorable resolutions. A complication in severe COVID-19 patients is the concurrent infection with other pathogens, particularly bacterial infections with multidrug-resistant strains. This work conducted a review to investigate the impact of the pandemic on healthcare systems in terms of the co-occurrence of SARS-CoV-2 infections and other bacterial pathogens, as well as the problem of antimicrobial resistance.
The results of this review show that co-infections mainly occur in patients with comorbidities and a more severe course of COVID-19. This implies a greater need for special care and prolonged hospital stays, increasing the possibility of complications and healthcare-associated infections. Initial indiscriminate antibiotic treatment practices by clinical staff were also reported, along with a shift in focus towards pandemic care, neglecting antimicrobial resistance surveillance programs.
In the antimicrobial resistance profiles, the presence of resistance mechanisms was evidenced, including an increase in the reporting of MRSA, Gram-negative bacilli with ESBL or NDM, with high resistance to carbapenems, polymyxin B, among others. Notable organisms include Acinetobacter. baumannii, Escherichia coli, Pseudomonas aeruginosa, Klebsiella pneumoniae, species of the genera Enterococcus and Streptococcus, Staphylococcus aureus including MRSA, among others.
Altogether, the need to continue monitoring, evaluating isolates, and establishing necessary protocols to provide appropriate management of co-occurrence of infections and antimicrobial resistance monitoring at local and regional levels is evident, especially in the face of large-scale epidemiological events such as the COVID-19 pandemic.
Keywords: COVID-19; Co-infections; Bacteria; SARS-CoV-2; Antimicrobial resistance.
Leer Artículo Completo
Introducción
El SARS-CoV-2 (Síndrome Respiratorio Agudo Grave y el tipo de virus Coronavirus 2) es el agente causante de la enfermedad denominada COVID-19 (“Coronavirus disease del 2019”)(1), considerada pandemia en el periodo 2020 – 2023. Al término de la declaración del fin de la pandemia en el 2023, se habían reportaron más de 676 millones de casos, con casi 7 millones de muertes (2).
La transmisión del virus se da a través de las gotas o aerosoles producidos por las personas infectadas cuando éstas son expelidas a través de la boca y/o nariz (3). Ante la exposición al agente, el virus ingresa a la célula utilizando el receptor de la enzima convertidora de la angiotensina (ACE 2) que se encuentra en las células humanas a nivel de pulmón. Con la infección consolidada, hasta un 80% de los casos pueden cursar como asintomáticos, y el resto presentan cuadros diversos con niveles que van desde leve a grave, siendo determinado no solo por las condiciones propias del individuo, sino que depende también de las variantes virales y el nivel previo de inmunización (4).
La presentación clínica se describe como variable, incluyendo diversos reportes iniciales asociados con fatiga, mialgias, cefalea y fiebre (3, 5), así como la ageusia o pérdida del gusto y la anosmia o pérdida del olfato (6). También, se han reportado síntomas digestivos como náuseas, vómitos y diarrea en un 10 a 20% de los casos (7). En cuadros graves el virus evade el sistema inmune e interfiere con la activación y producción de interferón, además, lleva a una gran migración de linfocitos y macrófagos que generan daño intersticial y desregulación de las citoquinas lo cual provoca el síndrome de hiperinflamación sistémica (denominada tormenta de citoquinas). Entre otros procesos fisiopatológicos, este complejo escenario termina por manifestar diversas condiciones clínicas, tales como síndrome de distrés respiratorio agudo (SDRA), insuficiencia respiratoria, miocarditis, daño renal agudo, falla orgánica múltiple, e incluso la muerte, en pacientes con casos graves de COVID-19 (8). Dentro de los escenarios de mayor complicación en el manejo de pacientes graves por COVID-19, se incluye la infección concomitante con otros patógenos, destacando las infecciones bacterianas incluso con cepas multirresistentes (MDR, por sus siglas en inglés) (9).
El objetivo de esta revisión fue obtener y estructurar la información disponible sobre los efectos de la pandemia por COVID-19 en la aparición de coinfecciones bacterianas y multirresistencias antimicrobianas mediante una revisión no sistemática de la literatura.
Metodología
Se realizó una revisión no sistemática de la literatura científica que incluía reportes de coinfecciones bacterianas en pacientes con SARS-CoV-2. Se realizó la búsqueda utilizando bases de datos como Google Académico, Medline (mediante el buscador PubMed), o Scielo, entre otras facilitadas por la Universidad de Costa Rica, para acceder a las fuentes de datos primarias para este estudio. Dentro de los criterios de inclusión se tuvo: (i) fecha de publicación a partir del año 2020; (ii) idioma inglés o español; (iii) palabras clave usadas (español e inglés): coinfección, SARS-CoV-2, COVID-19, sobreinfección, resistencia a antibióticos o antimicrobianos, bacterias, perfil de susceptibilidad o resistencia. Se sistematizó la información de cada artículo mediante el uso de un software automatizado para gestión de referencias Mendeley Reference Manager.
Síntesis de la revisión
Coinfección por SARS-CoV-2 y bacterias patógenas
La coinfección se define como la ocurrencia de al menos un segundo evento de infección por algún patógeno en condiciones de la coexistencia de una infección previa no resuelta (10) o por una infección múltiple al mismo tiempo (11). En este contexto, los estados de coinfección pueden llevar a dificultades a la hora de establecer el diagnóstico, el tratamiento y la prognosis de la enfermedad de COVID-19, aumentando los síntomas y la mortalidad (12).
Diferentes autores han descrito la ocurrencia de coinfecciones en pacientes con COVID-19, por diversos microorganismos como virus, hongos y bacterias. En parte, esto se debe a que el estado de infección por SARS-CoV-2 afecta la homeostasis de la microbiota y genera un ambiente favorable para desarrollar coinfecciones y posibles complicaciones, donde destacan infecciones secundarias por bacterias (12,13,14).
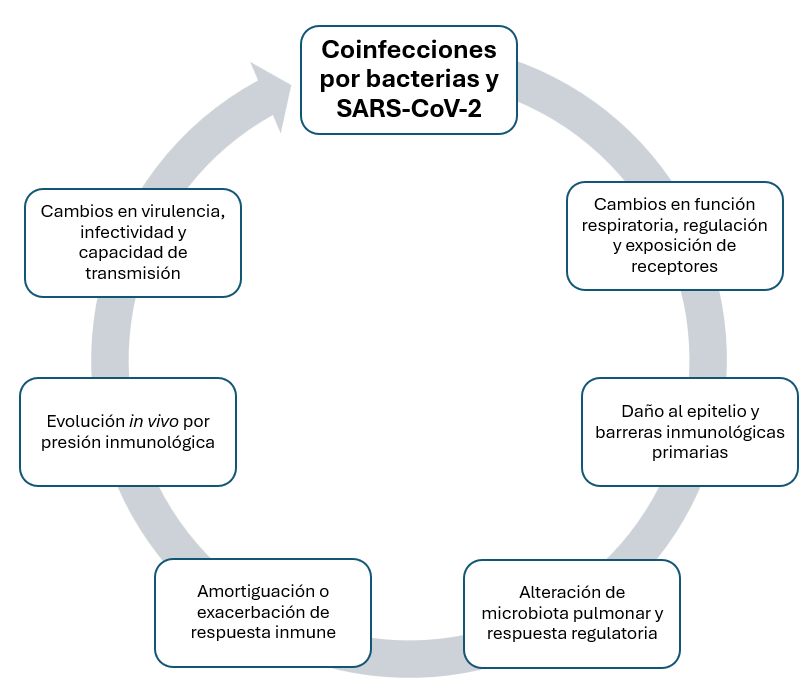
En este sentido, y como consecuencia de la evolución viral del SARS-CoV-2 que ocurre in vivo durante la infección en el hospedero, se puede afectar la virulencia, infectividad y capacidad de transmisión, debido a la presión inmunológica a la cual es sometido el virus y la posible interacción con otros microorganismos (15). También, la alteración en la microbiota pulmonar puede modificar la respuesta inmune y disparar la posibilidad de infecciones secundarias por bacterias (14). El entendimiento del papel que juega la microbiota puede ayudar a predecir el desarrollo de la coinfección y sus posibles complicaciones. Un resumen gráfico de estos aspectos clave se muestra en la Figura 1.
Figura 1. Factores clave en la ocurrencia de coinfecciones con SARS-CoV-2 y patógenos bacterianos.
Particularmente en coinfecciones con patógenos bacterianos, se describe una gran diversidad de agentes dependiendo de la epidemiología local, pero destacan: Acinetobacter baumannii, Escherichia coli, Pseudomonas aeruginosa, Enterococcus faecalis, Enterococcus faecium, Streptococcus pneumoniae, Staphylococcus aureus (incluyendo S. aureus resistente a la meticilina o MRSA), Haemophilus influenzae, Moraxella catarrhalis, Klebsiella pneumoniae y Enterobacter cloacae (11,16,17,18,19). Estos microorganismos no solo representan una seria complicación como causantes de infecciones nosocomiales, sino que regularmente se asocian a perfiles complejos de resistencia a los antibióticos.
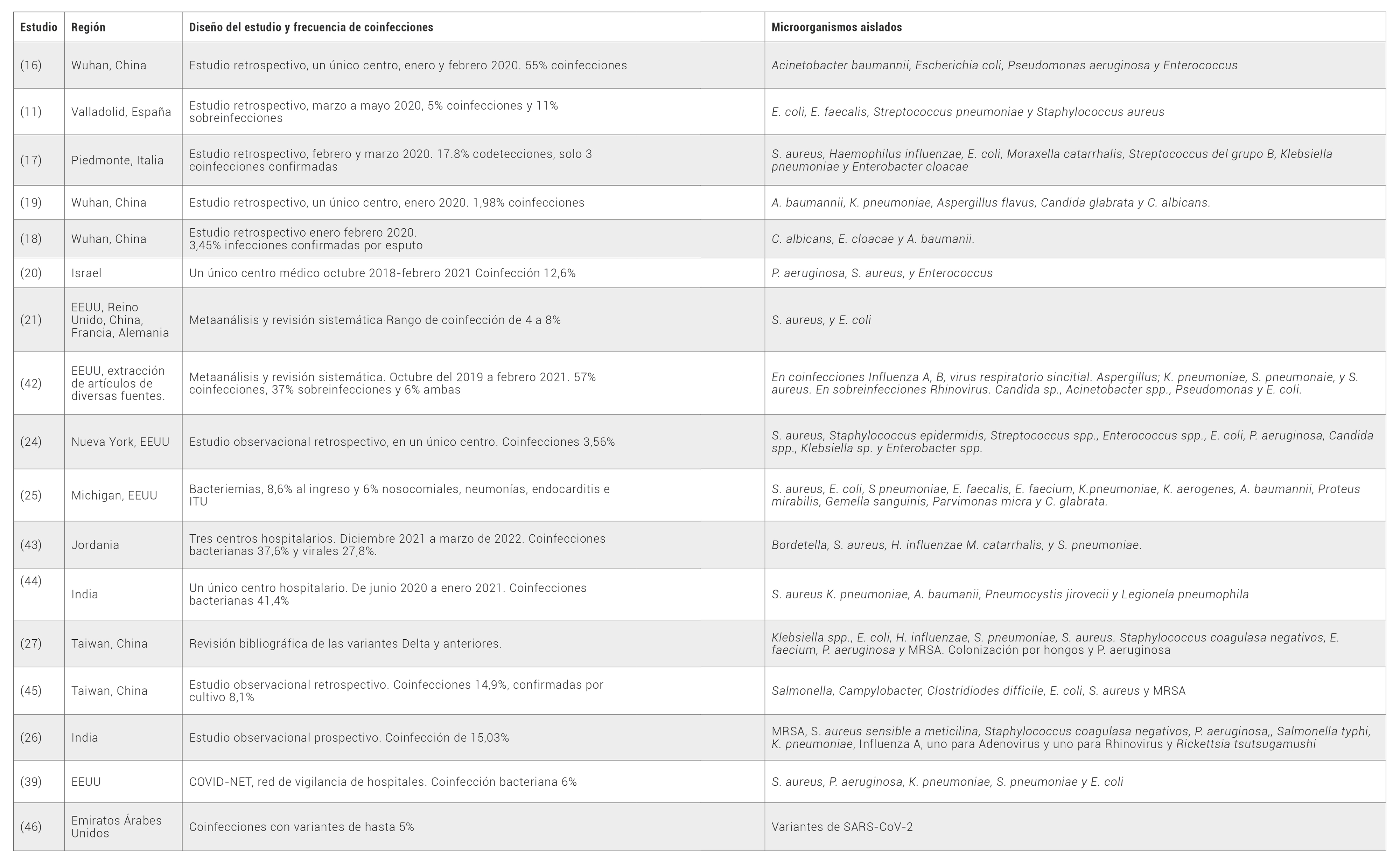
En la Tabla 1 se resumen los principales estudios de coinfección bacteriana en pacientes COVID-19, incluyendo lugar del estudio y principales aislamientos bacterianos registrados. En un estudio de Zhang et al. (16) se observó que las tasas de coinfecciones bacterianas eran significativamente más altas en aquellos pacientes con cuadros de COVID-19 graves. En el estudio se mencionan como razones principales la linfopenia y la inmunosupresión. Además, el hecho de que estos pacientes tengan dispositivos invasivos, como catéteres, incrementa la susceptibilidad de estos a infecciones bacterianas secundarias con patógenos nosocomiales multirresistentes como A. baumannii, E. coli, P. aeruginosa y Enterococcus.
Tabla 1. Resumen de los principales estudios sobre coinfecciones bacterianas en pacientes con COVID-19 seleccionados para esta revisión.
En el estudio retrospectivo realizado por Nebreda-Mayoral et al. (11) documentaron un 5% de coinfecciones principalmente de tracto urinario y respiratorias, dentro de los agentes etiológicos se encuentra E. coli, E. faecalis, S. pneumoniae y S. aureus, incluyendo casos que albergaban betalactamasas de espectro extendido (BLEE).
En un centro médico en Israel, se realizó un estudio en el cual se dividieron las infecciones secundarias bacterianas en tempranas y tardías, y se centraron en cultivos de esputo y sangre, comparando aquellos infectados con COVID-19 versus los infectados con virus influenza. Los marcadores asociados con inflamación y deterioro resultaron más elevados en pacientes con COVID-19, incluyendo proteína C reactiva, ferritina, lactato deshidrogenada y albúmina. Además, los pacientes COVID-19 tuvieron una mayor estadía hospitalaria, siete días versus cuatro días los de influenza, y tuvieron un mayor porcentaje de intubación 19,2% versus 4,4%. El porcentaje de infección bacteriana fue mayor en los infectados con COVID-19 12,6% versus 8,7% los de influenza, para el cual también presentaron una mayor tasa de mortalidad 26% versus 7%. En ambos grupos la mayoría de infecciones se dieron por bacterias Gram negativas, sin embargo, la cantidad de infecciones por Gram positivos fue mayor en los pacientes con COVID-19, 28% versus 9,5% (20).
Otro estudio, de un metaanálisis y una revisión sistemática realizado por Alshaikh et al. (21) incluyó estudios realizados en EEUU, Reino Unido, China, Francia, Alemania, entre otros, para analizar la prevalencia de las coinfecciones bacterianas en pacientes con COVID-19. La mayoría de los pacientes eran masculinos con una edad promedio de aproximadamente 61 años. Los patógenos más comunes fueron S. aureus y E. coli, y el principal foco fue pulmonar, luego sanguíneo y por último, el urinario. Los antibióticos más comúnmente usados fueron las cefalosporinas. La prevalencia de coinfecciones reportada al analizar los diferentes estudios fue de un 5,62%, con un uso de antibióticos en pacientes con COVID-19 que rondó un 61,16% de los casos. En cuanto a regiones, Norteamérica fue la que reportó un mayor porcentaje de coinfecciones con un 7,89%, Asia un 5,3% y Europa la más baja con un 3,57%. Con respecto al uso de antibióticos, la región de Norteamérica fue la que reportó un mayor uso con un 68,84%, Europa un 60,01% y Asia un 40,81% en los casos de COVID-19 con infección por bacterias.
Muchos estudios previos con muestras más pequeñas dan a conocer números similares a los obtenidos en el de Alshaikh et al. (21) con respecto a los porcentajes de coinfección bacteriana, un rango aproximado que va de 4% a 8%. Solo se mencionan tres estudios que poseen altas tasas de prevalencia de coinfecciones. Uno es el de Goncalves Mendes Neto et al. (22) que reportó un 19% de coinfecciones en que la mayoría corresponden a infecciones de tracto genitourinario (57%). En parte esto se debió a una definición laxa de coinfección, lo cual afecta también los microorganismos aislados, puesto que no se corresponden por ejemplo con los asociados a neumonía. El otro caso es el de Puzniak et al. (23) que reportó una prevalencia de coinfecciones del 16%, lo cual puede deberse a que la investigación sobre cultivos fue mucho mayor en este estudio que en otros.
Otro estudio realizado en un centro médico en Nueva York tomó en cuenta pacientes con cultivos respiratorios o hemocultivos positivos, y se compararon los datos de sensibilidad. El 60% tuvo cultivos respiratorios positivos, 54% hemocultivos positivos y un 14% ambos. Se reportó un 15% de bacterias multirresistentes, de estos un 5% fueron enterobacterias resistentes a carbapenémicos. En el caso de las coinfecciones evidenciadas en hemocultivos, la media de duración de positividad fue de 7 días, de estos 54% tenían catéter venoso central (24).
En una investigación realizada en Michigan, EEUU, se identificó la presencia de bacteriemias en 8,6% de los pacientes al momento de la admisión, mientras que se identificaron un 6% de nosocomiales. Dentro de los microorganismos aislados, se encontraron siete MRSA asociados a neumonía, endocarditis e infección de tracto urinario, dos E. coli BLEE, los E. faecium y E. faecalis resistentes a vancomicina. Un 65% de las bacteriemias en admisiones venían de asilos o centros de estancia con enfermerías, lo que podría explicar el alto porcentaje de MRSA (25).
En otro estudio prospectivo realizado en la India de junio a diciembre del 2020 en población pediátrica de un solo centro, se documentó una tasa de coinfección de 15,03% durante su admisión (26). La coinfección la definieron a través del aislamiento de un microorganismo en cultivo, un resultado positivo en paneles de PCR o estudios serológicos positivos. Dentro de las coinfecciones que se registraron 32,5% correspondieron a hemocultivos positivos, en los cuales se reportó 21,4% positivos por MRSA, 21,4% por S. aureus sensible a meticilina, 14,2% de Staphylococcus coagulasa negativos, 14,2% de P. aeruginosa, 7,14% de Salmonella typhi y 7,14% de K. pneumoniae. Solo tres pacientes presentaron crecimiento en líquido cefalorraquídeo, dos con MRSA y uno con S. aureus sensible a meticilina.
Existen otros estudios como los realizados por Wu et al. (27) en los que se documenta que la tasa de coinfección en pacientes ambulatorios es menor al 1%. Las tasas de coinfección de pacientes hospitalizados van de 2,5% a 5,1%. Sin embargo, podría existir una subestimación debido a como se clasifique la coinfección, sobre todo en aquellos estudios que usan cultivos, puesto que solo de 40 a 70% de los pacientes hospitalizados se hemocultivan y solo un 15 a 20% se les toman cultivos respiratorios. Otro dato importante es que se reportó una mayor colonización en los pacientes con COVID-19 por hongos y P. aeruginosa, pudiendo ésta última tener alguna incidencia sobre la generación de resistencia al tratamiento antimicrobiano.
En conjunto, a pesar de la variabilidad en los reportes anteriores, las coinfecciones de SARS-CoV-2 y bacterias, regularmente se reporta con agentes como S. aureus, especies del género Streptococcus, enterobacterias, P. aeruginosa y A. baumanii. Muchas de las diferencias que se observan entre los estudios pudieron deberse a los tipos de técnicas empleadas para determinar el estado de coinfección, en muchos casos PCR multiplex con variaciones en la cantidad y tipo de microorganismos detectados (28). Otros factores para tomar en cuenta son la edad de los grupos de estudio, si poseen enfermedades crónicas o factores de riesgo, su genética, tipo de tratamientos administrados, así como las características del agente viral. También, la realización de estudios para establecer como se afecta la severidad y mortalidad de los pacientes con COVID-19 y coinfecciones bacterianas es complicada en el sentido de que existen múltiples variables por definir: si la infección a considerar es temprana o tardía, el tipo de muestra o cultivo a tomar en cuenta, entre otros. En contraste, los estudios concuerdan sobre la relevancia de analizar al paciente para ver si posee un cuadro grave, y si existen signos y síntomas que sugieran una posible coinfección. Esto puede orientar al momento de solicitar las pruebas y cultivos correspondientes para poder diagnosticar adecuadamente el patógeno, determinar el perfil de susceptibilidad antimicrobiana y decidir sobre la terapia empírica de acuerdo con las guías de tratamiento instauradas con base en la epidemiología local.
Problema de resistencia a los antimicrobianos en el contexto de coinfecciones por patógenos bacterianos en pacientes COVID-19
La resistencia a los antimicrobianos es una seria amenaza a la salud humana, animal y ambiental, y requiere un abordaje multisectorial que incluyan decisiones sociales, políticas y económicas, dentro de una perspectiva que se denomina “una salud”. En mayo del 2015 la Organización Mundial de la Salud (OMS), aprobó el plan de acción global para la resistencia antimicrobiana, incluyendo la creación del sistema de vigilancia para el uso y resistencia de los antimicrobianos (GLASS). Con esta iniciativa se pretende patrocinar la vigilancia de la resistencia y proponer estrategias para contenerla, incluyendo expandir la vigilancia del consumo de antibióticos, y el modelo integrado de vigilancia de una salud (29).
Para el 2023 se emitió un reporte de 126 países participantes en el GLASS donde se mostraron las tasas de resistencia de las principales bacterias para el año 2020. Este reporte puso en evidencia inequidades en la cobertura y control de calidad de las pruebas, lo que se traduce en variaciones importantes en las tasas de resistencia entre países, sobre todo en aquellos de medianos y bajos ingresos económicos. A pesar de las brechas, logró identificar altas tasas de resistencia en los microorganismos asociados a infecciones del torrente sanguíneo, sobre todo para antibióticos de último recurso, como los carbapenémicos. Como se indica en el mismo documento, el diseño de nuevos antibióticos es muy caro y complejo, y muchas veces el desarrollo de resistencia a estos nuevos compuestos se presenta al poco tiempo de su uso, por lo que es de vital importancia tratar de mantenerlos factibles el mayor tiempo posible (29). Además de la afectación en la salud de las personas que quedan con muy limitadas opciones terapéuticas, las infecciones con microorganismos multirresistentes tienen un gran impacto económico, por ejemplo en EEUU se asocian con cerca de 100 mil muertes y un costo estimado de 4,5 a 11 mil millones de dólares por año (30).
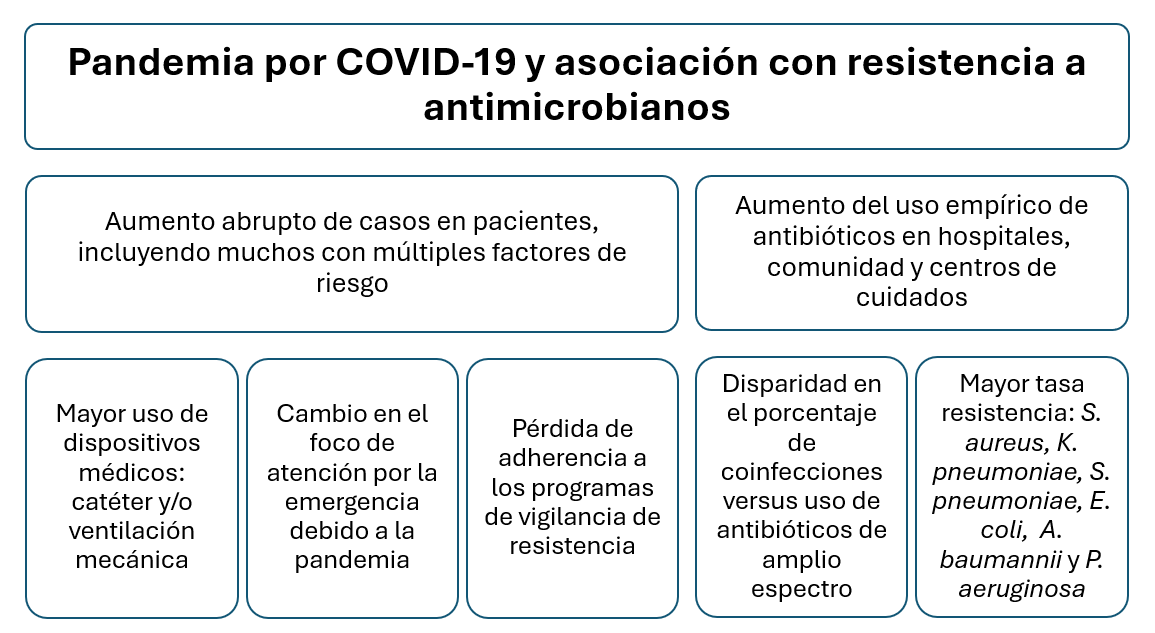
En el contexto de pandemia por COVID-19 alrededor del mundo, todos los sectores de la sociedad se vieron ampliamente afectados. A nivel salud, como se muestra en la Figura 2, se destacaron diferentes determinantes que impactaron el problema de resistencia a los antimicrobianos. Esa afectación no fue solo por el aumento de casos y la reducción en la disponibilidad de servicios, sino que también incluyó la reducción en los esfuerzos que se venían dando para vigilar la resistencia y el consumo de antimicrobianos a nivel hospitalario (31). Además, durante las primeras semanas de la pandemia en las que aún no se contaba con mucha información y no había disponibilidad de vacunas, el uso de antibióticos empíricos se disparó a nivel hospitalario, comunitario, así como en centros de cuidados para personas de la tercera edad o con situaciones especiales. Desde un punto de vista de manejo clínico, esto se hizo como una forma de evitar o tratar el aumento en las coinfecciones por agentes bacterianos o fúngicos en pacientes con COVID-19 grave, que presentaban además factores de riesgo y comorbilidades como la presencia de catéter venoso central, ventilación mecánica, uso de corticoesteroides, larga estancia hospitalaria, entre otros. En el Tabla 2 se resumen los principales estudios en los cuales se reportan coinfecciones con los microorganismos aislados y sus perfiles de resistencia asociados.
Figura 2. Determinantes del problema de resistencia a los antimicrobianos asociados a la ocurrencia de coinfecciones por patógenos bacterianos y SARS-CoV-2.
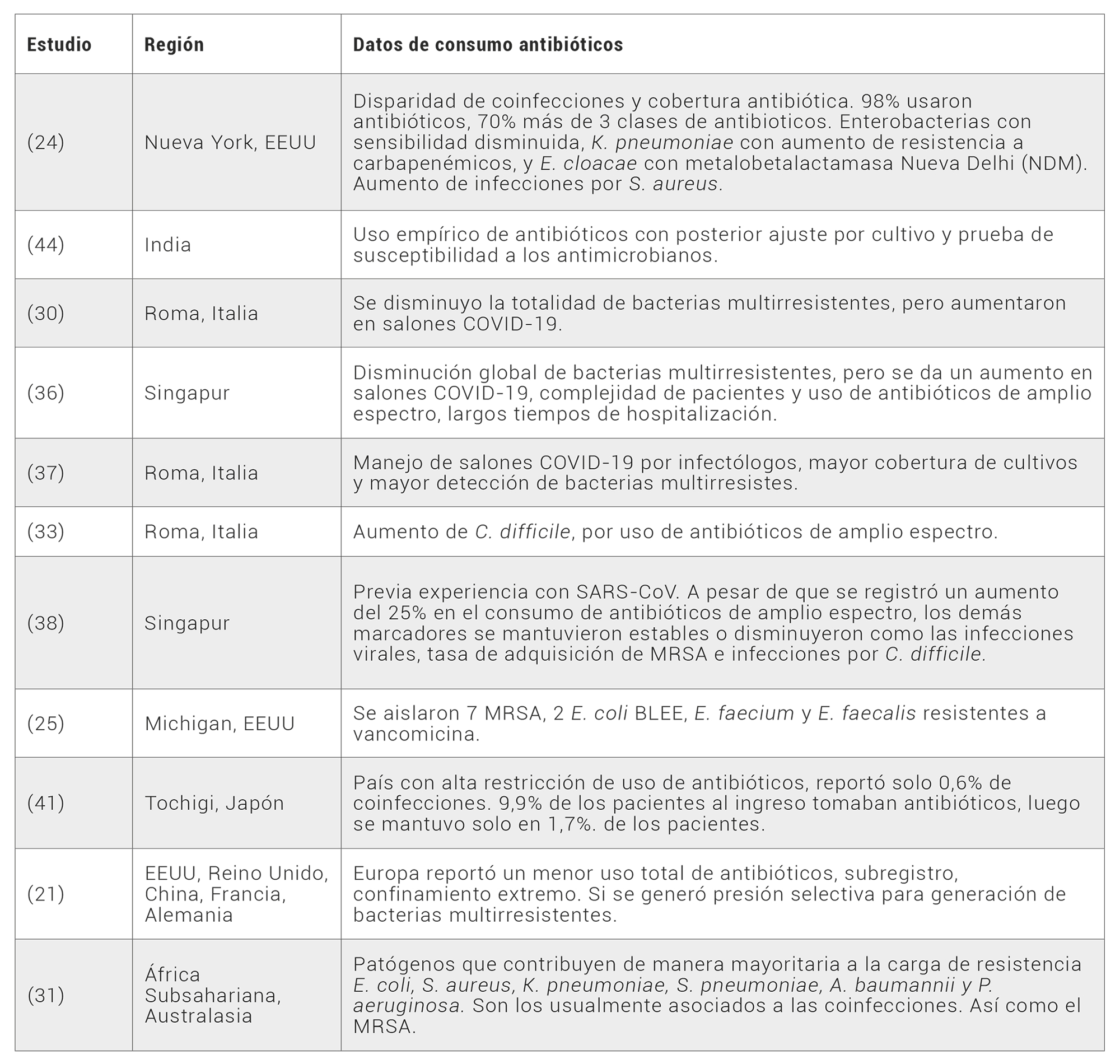
Tabla 2. Resumen de los estudios de coinfecciones con datos de resistencia a los antimicrobianos de los aislamientos reportados.
En un estudio realizado por Langford et al., (32), del análisis de 3506 pacientes se observó que el 71,8% recibieron antibióticos (principalmente quinolonas y cefalosporinas de tercera generación) pero solo un 3,5% fue diagnosticado con infección bacteriana durante la admisión, y de esos uno de cada siete eran infecciones asociadas a la atención en salud.
En otro estudio se reportó que las infecciones de tracto urinario eran las coinfecciones más comunes en pacientes provenientes de la comunidad, con un 57 a 70% de los casos y donde los principales patógenos asociados fueron E. coli y Klebsiella spp. (27) En el caso de infecciones respiratorias los patógenos más comunes reportados para coinfecciones adquiridas en la comunidad fueron H. influenzae, S. pneumoniae y S. aureus, y se trataron con cefalosporinas de tercera generación como ceftriaxona o con fluoroquinolonas (27). En otro reporte se documentó un aumento de los casos de Clostridioides difficile. Esto podría deberse al uso de antibióticos de amplio espectro que afectan la microbiota intestinal, o bien, consecuencia de que el mismo cuadro de COVID-19 generó una disbiosis en la microbiota de estos pacientes (33).
También, en otros estudios se ha reportado una incidencia más alta de patógenos multirresistentes en salones de COVID-19 que en otros servicios del hospital, 29% versus 19%, reflejado principalmente por K. pneumoniae con BLEE (30). En otro reporte, la prevalencia de coinfección con bacterias resistentes fue de 24%, y dentro de los microorganismos resistentes se encontraron MRSA, A. baumanii resistente a carbapenémicos, K. pneumoniae, y P. aeruginosa (34). Otros estudios revelaron que la tasa de resistencia de A. baumannii y K. pneumoniae a los carbapenémicos aumentó del 2019 al 2020, así como la resistencia de K. pneumoniae a la polimixina B de un 5% a un 50% (35).
Sin embargo, en otro trabajo se reportó una reducción inesperada de las bacterias multirresistentes en números generales, que podría deberse a la implementación universal de medidas de contención de la propagación de SARS-CoV-2 en todos los servicios. No obstante, cuando compararon los salones COVID-19 y otros salones, los primeros tuvieron una mayor incidencia de bacterias multirresistentes que los segundos. Esto podría ser contradictorio a la aseveración propuesta, pero los autores lo justificaron en una mayor complejidad de los pacientes COVID-19 y las terapias empíricas de antibióticos de amplio espectro que se solían usar mucho al inicio de la pandemia (36). Otro dato de relevancia podría ser el hecho de que en muchos salones COVID-19 la atención es manejada por médicos infectólogos, los cuales tienden a solicitar mayor cantidad de cultivos. Otra posible razón podría asociarse a las comorbilidades de los pacientes de COVID-19 que los llevaron a largos tiempos de hospitalización, permitiendo la colonización con bacterias multirresistentes (37).
En el estudio realizado en Singapur en los meses de febrero a abril 2020, se evaluaron las intervenciones realizadas por el equipo de optimización del uso de antimicrobianos con respecto al mismo periodo, pero del 2019. En este país se instauraron medidas bastantes estrictas en cuanto se generó la alarma, se declaró la pandemia y apareció el primer caso. Dentro de las medidas se incluyó el control de fronteras, cuarentena de contactos y viajeros a ciertas áreas, toma de temperatura y restricción de visitas al hospital. En los centros médicos se cancelaron los procedimientos electivos y no urgentes para tener mayor disponibilidad de camas y personal médico. A pesar de que la ocupación hospitalaria decreció un 16,8%, se incrementó en un 25,5% el uso de antibióticos de amplio espectro como cefepima, piperacilina-tazobactam, carbapenémicos y vancomicina, donde las dosis diarias definidas aumentaron de 14,92 por cada 100 días cama a 18,72, de similar manera ocurrió con las prescripciones para la neumonía asociada a la comunidad. El incremento fue más marcado al inicio de la pandemia en febrero 2020, sobre todo para ceftriaxona, amoxicilina con ácido clavulánico, levofloxacina, moxifloxacina, azitromicina y claritromicina (36). Al desconocer hasta cierto punto que se podía esperar con el SARS-CoV-2 y el desarrollo de la enfermedad de COVID-19, muchos de los servicios hospitalarios y su personal dejaron de lado el trabajo de rutina para avocarse completamente a la emergencia. En un principio se creyó que se podría contener la expansión y extensión del virus y se dejaron de lado programas de gran importancia epidemiológica. Sin embargo, con el tiempo se debió instaurar nuevamente las funciones del equipo del programa de optimización de los antimicrobianos (PROA), que al fin y al cabo generan un gran impacto en el manejo adecuado de las terapias antimicrobianas.
En otro estudio se evaluaron las consecuencias de la implementación de las medidas para el control y prevención de la infección por COVID-19 (mascarilla, la restricción de visitas e implementación de aislamiento por contacto) en las tasas de infecciones asociadas a la atención en salud. Al comparar las infecciones virales asociadas al cuidado de la salud, se redujeron de 9,69 a 0,83 casos por cada 10 000 pacientes. Las tasas de adquisición de MRSA disminuyeron al igual que las infecciones asociadas a catéter venoso central, además, no se registró aumento de las infecciones por C. difficile. Lo que si se observó fue un aumento importante del 25% en el consumo de antibióticos de amplio espectro como las cefalosporinas de cuarta generación, vancomicina y carbapenémicos (38).
Además, en el estudio Nori et al., (24) se mencionó que todos los pacientes con infecciones por bacterias multirresistentes tuvieron tratamientos previos con antibióticos. Además, realizaron una comparación de los consolidados microbiológicos en un periodo de dos meses de marzo a abril de 2019 y 2020. Encontraron que en el caso de los aislamientos de K. pneumoniae la susceptibilidad a las cefalosporinas, ciprofloxacina y meropenem bajó un 10%. Durante el primer mes de pandemia, se reportó a cinco pacientes con E. cloacae con metalobetalactamasa Nueva Delhi (NDM), en cultivos respiratorios y hemocultivos. Todos provenían de la comunidad, sin exposición a centros hospitalarios ni provenientes del extranjero y cuatro de los cinco murieron por shock séptico.
En el estudio realizado por Shah et al., (39) se reportó que el uso de antimicrobianos, sobre todo al principio de la pandemia era sumamente alto, de hasta 65% de prevalencia de su uso en los Estados Unidos (40).
En este estudio de Komagamine et al., (41), analizaron los pacientes hospitalizados en el centro médico Tochigi en Japón de noviembre del 2020 a octubre del 2021. De los 1056 pacientes, un 50% presentaron un cuadro leve, un 29,6% fue moderado, 19,2% fue grave y 0,7% crítico. Apenas un 9,9% de pacientes tenían prescripción de antibióticos antes del ingreso, pero solo el 1,7% siguió recibiendo antimicrobianos durante la hospitalización. Solo se confirmaron siete microorganismos coinfectando en seis pacientes, o sea un 0,6%. Esto sugirió que el hecho de no suministrar antibióticos de manera profiláctica no los hace más propensos a sufrir una coinfección.
Pese a la diversidad de reportes asociados a patógenos resistentes en el contexto de la pandemia por COVID-19, es importante considerar que en general el mayor consumo de antibióticos se da en países de medianos y bajos recursos, de manera similar a la distribución de los microrganismos multirresistentes. Esto quedó evidenciado en un estudio realizado de resistencia a los antimicrobianos, donde se estimaron 4,95 millones de infecciones con agentes resistentes que llevaron a la muerte, y de esos 1,27 millones de muertes son atribuibles directamente a la resistencia. Los patógenos que se asociaron a una mayor carga de resistencia fueron E. coli, S. aureus, K. pneumoniae, S. pneumoniae, A. baumannii y P. aeruginosa (31). En la lista de la OMS el MRSA está dentro de los de alta prioridad, pero aún no dentro de los de prioridad crítica. Esto podría cambiar dada la dinámica de la ocurrencia de infecciones concomitantes al SARS-CoV-2 (25), como se observó también durante la pandemia de influenza H1N1. Con este último virus, el MRSA adquirió gran relevancia al ser uno de los aislamientos más frecuentemente encontrados en los reportes de coinfecciones por neumonías sobre todo provenientes de la comunidad (31).
Basado en esta revisión, se pone en evidencia que la pandemia por COVID-19 ha sustentado la ya existente crisis de resistencia a los antimicrobianos desde distintos frentes, pero con el cese oficial del estado de emergencia y pandemia se han retomado las buenas prácticas en miras de contrarrestar los efectos de la misma, incluyendo: (i) retomar las medidas que eviten la prescripción sin supervisión e incremento en el uso de antibióticos de manera indiscriminada, (ii) reorganización de los programas de vigilancia como los PROA que permiten completar los informes de susceptibilidad a antimicrobianos, pese a la reasignación de personal a otros puestos durante el colapso de los sistemas de salud, y (iii) redireccionamiento de los recursos económicos de nuevo a la vigilancia de resistencia, que en su momento se desviaron para aumentar la infraestructura y recursos médicos para hacer frente a la pandemia de COVID-19.
Dentro de las limitaciones de este estudio, se evidenció una gran diversidad de condiciones y heterogeneidad de los trabajos analizados en cuanto a población, número de muestras, estrategias de selección y métodos diagnósticos, definiciones de lo que es coinfección, medidas de contención, periodo de tiempo, entre muchos otros. Tampoco se consideraron factores ambientales, medidas de protección y de contención, del huésped y de las variantes virales para las comparaciones. En el caso de Latinoamérica, existen escasos datos de coinfecciones y comparaciones de perfiles de resistencia tras la pandemia por COVID-19. En Costa Rica, a pesar de contar con un sistema de salud centralizado, no existen datos publicados sobre las coinfecciones que se dieron en los pacientes internados por COVID-19 a nivel de la seguridad pública.
Conclusiones
La pandemia por el agente SARS-CoV-2 tuvo un impacto en todos los ámbitos de la sociedad a nivel mundial. Pese a que aún se siguen estudiando las secuelas de la COVID-19, a nivel de salud han sido evidentes los cambios en los patrones de ocurrencia de infecciones concomitantes por bacterias multirresistentes a los antimicrobianos. Aun cuando existe una alta heterogeneidad en los estudios analizados, se coincide en revelar que las coinfecciones se presentan principalmente en pacientes con comorbilidades asociadas, que tienden a tener un cuadro más grave de COVID-19 y mayor necesidad de cuidados especiales que alargan su estancia hospitalaria y por ende a aumenta la posibilidad de complicaciones e infecciones asociadas al cuidado de la salud. También, las coinfecciones se relacionan con las prácticas iniciales de tratamiento indiscriminado con antibióticos por personal clínico, redirección a la atención de pandemia y la situación de emergencia sanitaria en general.
En los perfiles de resistencia antimicrobiana, se evidenció la presencia de mecanismos de resistencia importantes, incluyendo un aumento en el reporte de MRSA, bacilos Gram negativos con BLEE o con NDM, con alta resistencia a carbapenémicos, a la polimixina B, entre otros, que se reportan principalmente para A. baumannii, E. coli, P. aeruginosa, K. pneumoniae, especies de los géneros Enterococcus y Streptococcus, S. aureus MRSA, entre otros.
De esta forma, dada la circulación sostenida de SARS-CoV-2 pese a la declaratoria del fin de la pandemia, la fisiopatología de las infecciones virales y las características propias de los pacientes, la ocurrencia de coinfecciones con agentes bacterianos seguirá estando presente. Por ello es necesario seguir monitoreando, evaluar los aislamientos y establecer los protocolos necesarios para brindar un manejo adecuado de la problemática, en particular para cepas MDR. La implementación y revisión continúa de las guías de profilaxis con la epidemiología local actualizada tanto a nivel comunitario como hospitalario, son las que deben dirigir la posible implementación del tratamiento antimicrobiano.
A nivel individual, es imperativo mantener y extender medidas básicas de control de agentes infecciosos, como lavado de manos, aislamientos, desinfección de superficies e implementos, uso de mascarillas o equipo de protección, entre otros, mismos que podrían desde un inicio incidir en el éxito de la contención un posible brote. Desde la vigilancia de la resistencia a nivel local, también es necesario realizarla continuamente de forma preventiva (y no solo reactiva) en los PROA. Esto incluye la continua capacitación y concientización del personal sanitario respecto a las decisiones de tratamientos empíricos para infecciones bacterianas aún no confirmadas y su continuo seguimiento, así como la implementación de auditorías que permitan contener el uso excesivo o inadecuado de los antimicrobianos.
Financiación
Este trabajo se realizó con el apoyo de los proyectos “C0196 Protocolo bioinformático y de inteligencia artificial para el apoyo de la vigilancia epidemiológica basada en laboratorio del virus SARS-CoV-2 mediante la identificación de patrones genómicos y clínico-demográficos en Costa Rica (2020-2025), Vicerrectoría de Investigación, Universidad de Costa Rica (periodo 2021–2023) y “C4604 iPAT: Plataforma genómica, bioinformática y de inteligencia artificial para la vigilancia de patógenos”, Vicerrectoría de Investigación, Universidad de Costa Rica (periodo 2024–2026).
Referencias BIBLIOGRÁFICAS
- ↑Prieto R. Más allá de las pandemias – Beyond the pandemic. Rev colomb cir [Internet]. 2020;35(2):141–2. Available from: https://www.revistacirugia.org/index.php/cirugia/article/view/606/463 DOI: 10.30944/20117582.606
- ↑Coronavirus Resource Center. COVID-19 Map – Johns Hopkins Coronavirus Resource Center [Internet]. 2023. Available from: https://coronavirus.jhu.edu/map.html
- ↑Guan W jie, Ni Z yi, Hu Y, Liang W hua, Ou C quan, He J xing, et al. Clinical Characteristics of Coronavirus Disease 2019 in China. New England Journal of Medicine. 2020;382(18):1708–20. DOI: 10.1056/NEJMoa2002032
- ↑Dabanch J. Emergencia de SARS-COV-2. Aspectos básicos sobre su origen, epidemiología, estructura y patogenia para clínicos. Revista Clínica Las Condes [Internet]. 2021;32(1):14–9. DOI: 10.1016/j.rmclc.2020.12.003
- ↑Molina-Mora JA, González A, Jiménez-Morgan S, Cordero-Laurent E, Brenes H, Soto-Garita C, et al. Clinical Profiles at the Time of Diagnosis of SARS-CoV-2 Infection in Costa Rica During the Pre-vaccination Period Using a Machine Learning Approach. Phenomics 2022 [Internet]. 2022 Jun [cited 2022 Jun 7];1:1–11. DOI: 10.1101/2021.06.18.21259157
- ↑Lechien JR, Chiesa-Estomba CM, De Siati DR, Horoi M, Le Bon SD, Rodriguez A, et al. Olfactory and gustatory dysfunctions as a clinical presentation of mild-to-moderate forms of the coronavirus disease (COVID-19): a multicenter European study. European Archives of Oto-Rhino-Laryngology [Internet]. 2020; 277(8):2251–61. DOI: 10.1007/s00405-020-05965-1
- ↑Cheung KS, Hung IFN, Chan PPY, Lung KC, Tso E, Liu R, et al. Gastrointestinal Manifestations of SARS-CoV-2 Infection and Virus Load in Fecal Samples From a Hong Kong Cohort: Systematic Review and Meta-analysis. Gastroenterology. 2020,159(1):81–95. DOI: 10.1053/j.gastro.2020.03.065
- ↑Pérez A. G, Cordero R. C, Avendaño C. Fidel. Otro desafío de la naturaleza: el nuevo coronavirus virología y fisiopatología del SARS-COV-2. Vol. 15, Neumol Pediatr. 2020. DOI:10.51451/np.v15i2.69
- ↑Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. The Lancet. 2020,395(10223):497–506. DOI: 10.1016/S0140-6736(20)30183-5
- ↑Molina-Mora JA, Cordero-Laurent E, Calderón-Osorno M, Chacón-Ramírez E, Duarte-Martínez F. Metagenomic pipeline for identifying co-infections among distinct SARS-CoV-2 variants of concern: study cases from Alpha to Omicron. Sci Rep. 2022,12(1). DOI: 10.1038/s41598-022-13113-4
- ↑Nebreda-Mayoral T, Miguel-Gómez MA, March-Rosselló GA, Puente-Fuertes L, Cantón-Benito E, Martínez-García AM, et al. Bacterial/fungal infection in hospitalized patients with COVID-19 in a tertiary hospital in the Community of Castilla y León, Spain. Enferm Infecc Microbiol Clin. 2022,40(4):158–65. DOI: 10.1016/j.eimce.2022.02.002
- ↑Shen Z, Xiao Y, Kang L, Ma W, Shi L, Zhang L, et al. Genomic diversity of severe acute respiratory syndrome-coronavirus 2 in patients with coronavirus disease 2019. Clinical Infectious Diseases. 2020;71(15):713–20. DOI: 10.1093/cid/ciaa203
- ↑Guo T, Fan Y, Chen M, Wu X, Zhang L, He T, et al. Cardiovascular Implications of Fatal Outcomes of Patients with Coronavirus Disease 2019 (COVID-19). JAMA Cardiol. 2020;5(7):811–8. DOI: 10.1001/jamacardio.2020.1017
- ↑Hanada S, Pirzadeh M, Carver KY, Deng JC. Respiratory viral infection-induced microbiome alterations and secondary bacterial pneumonia. Front Immunol. 2018; 9:26409:1–15. DOI: 10.3389/fimmu.2018.02640
- ↑Molina-Mora JA. Insights into the mutation T1117I in the spike and the lineage B.1.1.389 of SARS-CoV-2 circulating in Costa Rica. Gene Rep [Internet]. 2022 [cited 2022 Feb 8];27:1–9. Available from: https://linkinghub.elsevier.com/retrieve/pii/S2452014422000620 DOI: 10.1038/s41598-022-13113-4
- ↑Zhang G, Hu C, Luo L, Fang F, Chen Y, Li J, et al. Clinical features and short-term outcomes of 221 patients with COVID-19 in Wuhan, China. Journal of Clinical Virology [Internet]. 2020;127:104364. DOI: 10.1016/j.jcv.2020.104364
- ↑Calcagno A, Ghisetti V, Burdino E, Trunfio M, Allice T, Boglione L, et al. Co-infection with other respiratory pathogens in COVID-19 patients. Clinical Microbiology and Infection. 2021;27(2):297–8. DOI: 10.1016/j.cmi.2020.08.012
- ↑Wang Z, Yang B, Li Q, Wen L, Zhang R. Clinical features of 69 cases with coronavirus disease 2019 in Wuhan, China. Clinical Infectious Diseases. 2020;71(15):769–77. DOI: 10.1093/cid/ciaa272
- ↑Chen N, Zhou M, Dong X, Qu J, Gong F, Han Y, et al. Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study. The Lancet. 2020;395(10223):507–13. DOI: 10.1016/S0140-6736(20)30211-7
- ↑Shafran N, Shafran I, Ben-Zvi H, Sofer S, Sheena L, Krause I, et al. Secondary bacterial infection in COVID-19 patients is a stronger predictor for death compared to influenza patients. Nature Portfolio Scientific Reports [Internet]. 2021;11(123):12703. DOI: 10.1038/s41598-021-92220-0
- ↑Alshaikh FS, Godman B, Sindi ON, Andrew Seaton R, Kurdi A. Prevalence of bacterial coinfection and patterns of antibiotics prescribing in patients with COVID-19: A systematic review and metaanalysis. Vol. 17, PLoS ONE. Public Library of Science; 2022. DOI: 10.1371/journal.pone.0272375
- ↑Goncalves Mendes Neto A, Lo KB, Wattoo A, Salacup G, Pelayo J, DeJoy R, et al. Bacterial infections and patterns of antibiotic use in patients with COVID-19. J Med Virol. 2021;93(3):1489–95. DOI: 10.1002/jmv.26441
- ↑Puzniak L, Finelli L, Yu KC, Bauer KA, Moise P, De Anda C, et al. A multicenter analysis of the clinical microbiology and antimicrobial usage in hospitalized patients in the US with or without COVID-19. BMC Infect Dis. 2021;21(1). DOI: 10.1186/s12879-021-05877-3
- ↑Nori P, Cowman K, Chen V, Bartash R, Szymczak W, Madaline T, et al. Bacterial and fungal coinfections in COVID-19 patients hospitalized during the New York City pandemic surge. Infect Control Hosp Epidemiol. 2021;42(1):84–8. DOI: 10.1017/ice.2020.368
- ↑Bhargava A, Riederer K, Sharma M, Fukushima EA, Johnson L, Saravolatz L. High rate of Multidrug-Resistant Organisms (MDROs) among COVID-19 patients presenting with bacteremia upon hospital admission. Am J Infect Control. 2021;49(11):1441–2. DOI: 10.1016/j.ajic.2021.08.010
- ↑Raychaudhuri D, Sarkar M, Roy A, Roy D, Datta K, Sengupta T, et al. COVID-19 and Co-infection in children: The Indian perspectives. Vol. 67, Journal of Tropical Pediatrics. Oxford University Press; 2021. DOI: 10.1093/tropej/fmab073
- ↑Wu HY, Chang PH, Chen KY, Lin IF, Hsih WH, Tsai WL, et al. Coronavirus disease 2019 (COVID-19) associated bacterial coinfection: Incidence, diagnosis and treatment. Vol. 55, Journal of Microbiology, Immunology and Infection. Elsevier Ltd; 2022. p. 985–92. DOI: 10.1016/j.jmii.2022.09.006
- ↑Roh KH, Kim YK, Kim SW, Kang ER, Yang YJ, Jung SK, et al. Coinfections with Respiratory Pathogens among COVID-19 Patients in Korea. Canadian Journal of Infectious Diseases and Medical Microbiology. 2021;2021. DOI: 10.1155/2021/6651045
- ↑Global Antimicrobial Resistance and Use Surveillance System (GLASS) Report 2022 [Internet]. Vol. CC BY-NC-SA 3.0 IGO. 2022. Available from: https://www.who.int/publications/book-orders.
- ↑Bentivegna E, Luciani M, Arcari L, Santino I, Simmaco M, Martelletti P, et al. Reduction of Multidrug-Resistant (MDR) Bacterial Infections during the COVID-19 Pandemic: A Retrospective Study. J Environ Res Public Health [Internet]. 2021;18. DOI: 10.3390/ijerph18031003
- ↑Murray CJ, Ikuta KS, Sharara F, Swetschinski L, Robles Aguilar G, Gray A, et al. Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis. The Lancet. 2022;399(10325):629–55. DOI: 10.1016/S0140-6736(21)02724-0
- ↑Langford BJ, So M, Raybardhan S, Leung V, Westwood D, MacFadden DR, et al. Bacterial co-infection and secondary infection in patients with COVID-19: a living rapid review and meta-analysis. Vol. 26, Clinical Microbiology and Infection. Elsevier B.V.; 2020. p. 1622–9. DOI: 10.1016/j.cmi.2020.07.016
- ↑Bentivegna E, Alessio G, Spuntarelli V, Luciani M, Santino I, Simmaco M, et al. Impact of COVID-19 prevention measures on risk of health care-associated Clostridium difficile infection. Am J Infect Control. 2021;49(5):640–2. DOI: 1016/j.ajic.2020.09.010
- ↑Kariyawasam RM, Julien DA, Jelinski DC, Larose SL, Rennert-May E, Conly JM, et al. Antimicrobial resistance (AMR) in COVID-19 patients: a systematic review and meta-analysis (November 2019–June 2021). Antimicrob Resist Infect Control. 2022;11(1). DOI: 10.1186/s13756-022-01085-z
- ↑Gaspar GG, Ferreira LR, Feliciano CS, Campos Júnior CP, Molina FMR, Vendruscolo ACS, et al. Pre-and post-covid-19 evaluation of antimicrobial susceptibility for healthcare-associated infections in the intensive care unit of a tertiary hospital. Rev Soc Bras Med Trop. 2021;54. DOI: 10.1590/0037-8682-0090-2021
- ↑Liew Y, Lee WHL, Tan L, Kwa ALH, Thien SY, Cherng BPZ, et al. Antimicrobial stewardship programme: a vital resource for hospitals during the global outbreak of coronavirus disease 2019 (COVID-19). Int J Antimicrob Agents. 2020;56(5). DOI: 10.1016/j.ijantimicag.2020.106145
- ↑Luciani M, Bentivegna E, Spuntarelli V, Lamberti PA, Cacioli G, Del Porto F, et al. Recurrent COVID-19 pneumonia in the course of chemotherapy: Consequence of a weakened immune system? Vol. 93, Journal of Medical Virology. John Wiley and Sons Inc; 2021. p. 1882–4. DOI: 10.1002/jmv.26701
- ↑Wee LEI, Conceicao EP, Tan JY, Magesparan KD, Amin IBM, Ismail BBS, et al. Unintended consequences of infection prevention and control measures during COVID-19 pandemic. Am J Infect Control. 2021;49(4):469–77. DOI: 10.1016/j.ajic.2020.10.019
- ↑hah MM, Patel K, Milucky J, Taylor CA, Reingold A, Armistead I, et al. Bacterial and viral infections among adults hospitalized with COVID-19, COVID-NET, 14 states, March 2020-April 2022. Influenza Other Respir Viruses. 2023;17(3):e13107. DOI: 10.1111/irv.13107
- ↑Langford BJ, So M, Raybardhan S, Leung V, Soucy J paul R, Westwood D, et al. Antibiotic prescribing in patients with COVID-19 : rapid review and. Clinical Microbiology and Infection [Internet]. 2021;27(4):520–31. DOI: 10.1016/j.cmi.2020.12.018
- ↑Komagamine J, Yabuki T, Matsumoto K, Tanaka N. Evaluation of Antimicrobial Drug Use and Concurrent Infections during Hospitalization of Patients with COVID-19 in Japan. JAMA Netw Open. 2022;5(2). DOI: 10.1001/jamanetworkopen.2022.0040
- Musuuza JS, Watson L, Parmasad V, Putman-Buehler N, Christensen L, Safdar N. Prevalence and outcomes of co-infection and superinfection with SARS-CoV-2 and other pathogens: A systematic review and metaanalysis. PLoS One. 2021;16. DOI: 10.1371/journal.pone.0251170
- Khasawneh AI, Himsawi NM, Abu-Raideh JA, Sammour A, Abu Safieh H, Obeidat A, et al. Prevalence of SARS-COV-2 and other respiratory pathogens among a Jordanian subpopulation during Delta-to-Omicron transition: Winter 2021/2022. Liu BM, editor. PLoS One [Internet]. 2023 Mar 30 [cited 2023 May 5];18(3):e0283804. Available from: https://dx.plos.org/10.1371/journal.pone.0283804 DOI: 10.1371/journal.pone.0283804
- Sreenath K, Batra P, Vinayaraj E V., Bhatia R, SaiKiran K, Singh V, et al. Coinfections with Other Respiratory Pathogens among Patients with COVID-19. Microbiol Spectr. 2021;9(1). DOI: 10.1128/Spectrum.00163-21
- Lai HC, Hsu YL, Lin CH, Wei HM, Chen JA, Low YY, et al. Bacterial coinfections in hospitalized children with COVID-19 during the SARS-CoV-2 Omicron BA.2 variant pandemic in Taiwan. Front Med (Lausanne). 2023;10. DOI: 10.3389/fmed.2023.1178041
- Liu R, Wu P, Ogrodzki P, Mahmoud S, Liang K, Liu P, et al. Genomic epidemiology of SARS-CoV-2 in the UAE reveals novel virus mutation, patterns of co-infection and tissue specific host immune response. Sci Rep. 2021;11(1). DOI: 10.1038/s41598-021-92851-3
INFORMACIÓN DEL ARTÍCULO
Conflicto de intereses: Los autores de este artículo declaran no tener ningún tipo de conflicto de intereses respecto a lo expuesto en el presente trabajo.
Correspondencia: Jose Arturo Molina Mora. Universidad de Costa Rica, San José, Costa Rica. E-mail: jose.molinamora@ucr.ac.cr