Cartas al Editor
Desarrollo de nuevos métodos de detección para SARS-CoV-2 basados en la tecnología CRISPR
Development of new detection methods for SARS-CoV-2 based on CRISPR technology
Actual Med. 2020; 105(811): 244-245 DOI: 10.15568/am.2020.811.cd01
Recibido: 11/06/2020
Revisado: 23/06/2020
Aceptado: 11/07/2020
Leer Artículo Completo
A finales de diciembre del 2019 apareció una nueva neumonía inducida por un coronavirus con un brote inusual en Wuhan (China). La enfermedad es causada por el coronavirus 2 del síndrome respiratorio agudo severo (SARS-CoV-2) y fue nombrada COVID-19 por la OMS el 11 de febrero de 2020, el 11 de marzo de 2020 la OMS consideró que podía ser considerada una pandemia (1). El SARS-CoV-2 es un virus envuelto con un genoma de ARN monocatenario, que inicia la infección por el reconocimiento del receptor ACE2 (enzima convertidora de angiotensina 2) de células humanas (2).
El método más frecuente para detectar ácidos nucleicos es la reacción en cadena de la polimerasa (PCR), esta es capaz de detectar <100 copias de ácido nucleico por muestra, requiere equipamiento caro y especializado, y si la concentración de ácidos nucleicos es baja, puede no ser suficientemente sensible (3). Por lo que se han desarrollado otras técnicas basadas en la tecnología CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) más sensibles y específicas, como son SHERLOCK, DETECTR o CARMEN.
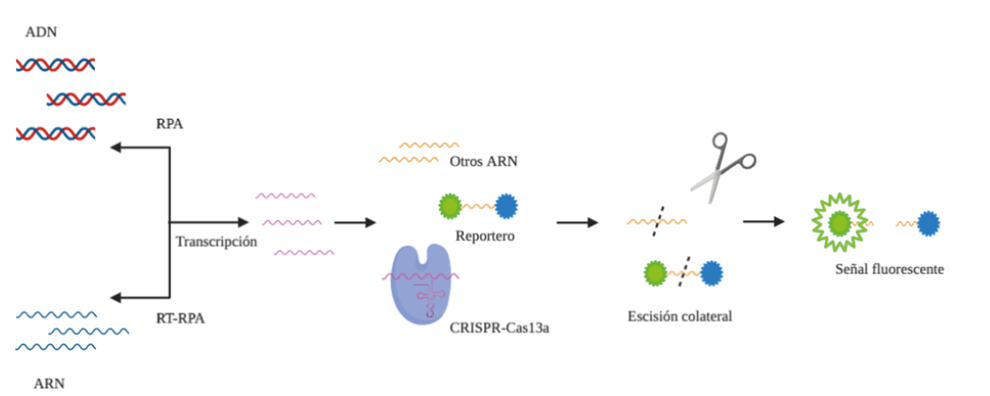
La técnica SHERLOCK (Specific High-sensitivity Enzymatic Reporter unLOCKing) combina la amplificación isotérmica (60-65ºC) de los ácidos nucleicos con la tecnología CRISPR. La endonucleasa utilizada es Cas13a que detecta ARN o ADN (utilizando una transcriptasa inversa) específico produciendo un fenómeno denominado “escisión colateral” en el cual se producen cortes aleatorios de secuencia de ARN(3). Tras la amplificación isotérmica se combinarían los fragmentos con CRISPR/Cas13a y ARNs reporteros, de forma que cuando CRISPR/Cas13a reconociera la secuencia de homología se escindirían fragmentos de ARN, entre ellos, algunos de los reporteros, con lo que se emitiría fluorescencia (3). Se ha comprobado su eficiencia en la detección de enfermedades infecciosas (4), pero no hay estudios que corroboren su eficacia para la detección del CoV-Sars-2.
La técnica DETECTR se basa en el mismo fundamento que el de la plataforma SHERLOCK, emplea la endonucleasa Cas12a que a diferencia de la Cas13a reconoce secuencias de ADN(5). Al tratarse de un virus de ARN, la amplificación isotérmica requiere de la realización de una transcripción inversa (RT-LAMP). Aa pesar de que recientemente ha sido presentado el desarrollo y validación inicial de un ensayo basado en CRISPR-Cas12 para la detección del SARS-CoV-2 a partir de ARN extraído de muestras de pacientes (5).
Finalmente, la técnica CARMEN (Combinatorial Arrayed Reactions for Multiplexed Evaluation of Nucleic acid) combina la tecnología de microfluídica con los métodos de detección mencionados, resultando en una técnica de diagnóstico escalable a cientos de muestras. El desarrollo de la plataforma CARMEN permite la detección de patógenos de forma multiplicada y adaptable a un mayor número de muestras (6). Utiliza la detección basada en Cas13 con ARN guía específico, las muestras amplificadas y reporteros marcados con fluorocromos que sirven como identificadores ópticos. CARMEN-Cas13 ha demostrado ser eficaz para el SARS-CoV-2 (6).
En definitiva, se han desarrollado nuevas técnicas más eficientes y específicas para la detección de ácidos nucleicos en muestras humanas con la finalidad de detectar de una forma más rápida y sencilla patógenos que puedan causar enfermedades, aun así, se precisa de más evidencias para la verificación de su efectividad en la detección del SARS-CoV-2.
Figura 1. Plataforma de diagnóstico SHERLOCK para reconocimiento específico de secuencias de ADN o ARN basada la combinación de la amplificación isotérmica de ácido nucleico con la enzimología CRISPR-Cas13a. RPA: amplificación recombinasa polimerasa. RT-RPA: transcriptasa reversa en amplificación recombinasa polimerasa. Elaboración propia basada en Gootenberg, 2017 (4).
AGRADECIMIENTOS
Nos gustaría agradecer a la Universidad de Murcia la posibilidad de formarnos, al Máster de Biología y Tecnología de la Reproducción en Mamíferos por acogernos y sobre todo a nuestro profesor el Dr. Joaquín Gadea por enseñarnos a aprender y por animarnos a descubrir el mundo de la ciencia.
REFERENCIAS BIBLIOGRÁFICAS
- ↑Cucinotta D, Vanelli M. WHO Declares COVID-19 a Pandemic. Acta biolomedica Atenei Parm [Internet]. 2020;91(1):157–60. DOI: 10.23750/abm.v91i1.9397
- ↑He F, Deng Y, Li W. Coronavirus disease 2019: What we know? J Med Virol. 2020;(March):1–7. DOI: 10.1002/jmv.25766
- ↑Gronowski A. Who or what is SHERLOCK? Int Fed Clin Chem Lab Med. 2018;29(3):1–26.
- ↑Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science (80). 2017;356(6336):438–42. DOI: 10.1126/science.aam9321
- ↑Broughton JP, Deng X, Yu G, Fasching CL, Servellita V, Singh J, et al. CRISPR – Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 2020;1–5. DOI: 10.1038/s41587-020-0513-4
- ↑Ackerman CM, Myhrvold C, Gowtham Thakku S, Freije CA, Metsky HC, Yang DK, et al. Massively multiplexed nucleic acid detection with Cas13. Nature. 2020; 582 (7811): 277-282. DOI: 10.1038/s41586-020-2279-8
INFORMACIÓN DEL ARTÍCULO
Conflicto de intereses: Los autores/as de este artículo declaran no tener ningún tipo de conflicto de intereses respecto a lo expuesto en el presente trabajo.
Autor para la correspondencia: Balma García-Colomer. Máster en Biología y Tecnología de la Reproducción en Mamíferos. Universidad de Murcia. E-mail: balma.garciac@um.es