Original
Sepsityper® para la identificación rápida de microorganismos a partir de hemocultivos positivos
Sepsityper® for rapid identification of microorganisms from positive blood cultures
Actual. Med. 2014; 99: (792): 70-74 DOI: 10.15568/am.2014.792.or03
Enviado 06-05-2014
Revisado 20-08-2014
Aceptado 26-08-2014
RESUMEN
Nuestro estudio ha evaluado las ventajas de la utilización del kit Sepsityper® para la identificación rápida de microorganismos a partir de hemocultivos positivos, acompañado de la tecnología de espectrometría de masas MALDITOF MS®, en comparación con los métodos tradicionales empleados para el diagnóstico de bacteriemia. Para la identificación del microorganismo en 379 hemocultivos positivos en el Departamento de Microbiología del Hospital Universitario San Cecilio, se aplicó la espectrometría de masas MALDITOF MS® utilizando el sistema Sepsityper® (Bruker) y se comparó con la identificación mediante métodos convencionales (Wider, Vitek II, Api). La correlación de resultados de los dos esquemas diagnósticos fue determinada estadísticamente por el coeficiente de correlación kappa. La distribución de los aislamientos fue de un 24,7 % de Bacilos Gram negativos (BGN) y 75,3 % de microorganismos Cocos Gram positivos (CGP). La concordancia global de resultados fue del 95,8 % en la especie (k = 0,928) y del 98,7 % en el género (k = 0,977), siendo el porcentaje de identificaciones fallidas del 1,3%. Para BGN hubo una concordancia de resultados del 95,2 % (k = 0,928, especie), y 100 % (k = 1, género). Respecto a los CGP, la concordancia fue del 98,2 % en género (k = 0,931), y del 82,5 % (k = 0,627) a nivel de especie. En nuestra experiencia se ha observado una ganancia de al menos 13–23h en la identificación a nivel de especie. La utilización del kit Sepsityper® para la identificación rápida de microorganismos a partir de hemocultivos positivos, acompañado de MALDITOF MS®, muestra una excelente correlación respecto a la identificación realizada a través de la metodología convencional, con una importante disminución del tiempo hasta la identificación.
Palabras clave: Malditof-MS®, bacteriemia, sepsityper®, hemocultivo, implementación
ABSTRACT
Our study has evaluated the advantages of using Sepsityper® kit for a fast identification of microorganisms from positive blood cultures, along with the mass spectrometry technology MALDITOF-MS®, compared to traditional methods used for diagnosis of bacteremia. To identify the microorganism isolated from the 379 positive blood cultures (BC +) the Department of Microbiology, University Hospital San Cecilio, MALDITOF-MS® mass spectrometry along with Sepsityper® (Bruker) were applied and it was compared to the conventional methods for the identification of this organism. The correlation of results between these two diagnostic schemes was statistically determined by kappa correlation coefficient. The distribution of the isolates was 24.7% for Gram negative bacilli (GNB) and 75.3% for Gram-positive cocci (GPC). The overall concordance of results was 95.8% within the species (k = 0.928) and 98.7% within the genus (k = 0.977), with a failed-identification percentage of 1.3%. For GNB there was a concordance of results of 95.2% (k = 0.928, species), and 100% (k = 1, genus). Regarding the GPC, the concordance was 98.2% within the genus (k = 0.931), and 82.5% (k = 0.627) at the species level. According to our experience there was a gain of at least 13-23 hours in the identification of the microorganisms at the species level. The use of Sepsityper® kit for the rapid identification of microorganisms from positive blood cultures, along with MALDITOF-MS®, show an excellent correlation compared to identification made by the conventional methods, with a significant reduction in time until identification.
Keywords: Malditof-MS®, bacteriemia, sepsityper®, blood cultures, implementation
Leer Artículo Completo
Introducción
La mortalidad del paciente con sepsis asociada a bacteriemia está directamente asociada con la implantación de un tratamiento antibiótico empírico inadecuado (1). Se estima que entre un 28 y un 34% de tratamientos empíricos que se instauran para el tratamiento de una sepsis son inadecuadas (2-4). Por ello, se necesitan esquemas terapéuticos empíricos optimizados para contribuir a evitar la aparición y diseminación de resistencias microbianas debidas a un uso inadecuado de antimicrobianos en diversas situaciones clínicas, así como mejoras en la rapidez y calidad del diagnóstico etiológico de bacteriemia. Entre las innovaciones metodológicas, la espectrometría de masas permite hacer un diagnóstico etiológico rápido en la rutina asistencial para el diagnóstico de la bacteriemia.
La espectrometría de masas ha sido utilizada en distintos aspectos relacionados con la identificación bacteriana desde 1980 (5-9), pero a partir de 2009, el sistema MALDI-TOF MS® (matrix assisted laser desorption ionization time-of-flight) comienza a utilizarse para el diagnóstico microbiológico, gracias a un protocolo de identificación de microorganismos basado en su perfil proteómico. Diversos autores han comparado esta tecnología tanto con los métodos tradicionales del laboratorio de microbiología (10-11), como con equipos basados en reacción de la cadena polimerasa a tiempo real tales como LightCycler SeptiFast, Roche Molecular Systems (PCR) (12-13). Frente a las pruebas bioquímicas, la rapidez es la gran ventaja de la tecnología de MALDI-TOF, mientras que frente a los métodos rápidos de PCR, la menor laboriosidad permite mejorar los flujos de trabajo en los laboratorios asistenciales, y su menor coste económico permite mejoras en las medidas de contención del gasto.
La aplicación de esta tecnología al estudio de los hemocultivos permite hacer el diagnóstico etiológico de la bacteriemia en minutos a partir del momento en que se positivizan los frascos, permitiendo por tanto hacer un tratamiento antibiótico dirigido a través de la identificación del microorganismo causal, con el consiguiente beneficio para el paciente.
En nuestro estudio presentamos los resultados de la implementación de un esquema de diagnóstico etiológico rápido de bacteriemia basado en la utilización del kit Sepsityper® seguido de la espectrometría de masas MALDITOF-MS®, para la identificación rápida de microorganismos a partir de hemocultivos positivos, determinando el grado de concordancia de resultados respecto a los métodos clásicos de diagnóstico microbiológico del agente etiológico de bacteriemia, y el beneficio en tiempo de adelanto que ofrece esta tecnología a la hora de permitir establecer un tratamiento antibiótico adecuado en un paciente séptico grave.
Material y métodos
Durante un periodo de tiempo de diez meses se compararon los resultados de 379 frascos de hemocultivos (BacT/ALERT®, Biomérieaux) con crecimiento bacteriano positivo correspondientes a 166 pacientes hospitalizados en el Hospital Universitario San Cecilio. Los frascos de hemocultivo positivos procedían de distintas tomas de pacientes (en condiciones atmosféricas de aerobiosis y anaerobiosis) durante la evolución clínica del paciente.
La distribución de los pacientes fue la siguiente: 15 pacientes pediátricos y 151 pacientes adultos (90.96% de la muestra), de los cuales el 64.45% fueron mujeres (107/166), con una mediana de edad de 55 años (37-71). La totalidad de las muestras pediátricas procedieron de la Unidad de Cuidados Intensivos Pediátricos del hospital, mientras que las 151 muestras de pacientes adultos analizadas, tuvieron la siguiente distribución por Servicios: 44.37% procedieron del área de urgencias externas (67/151); 54/151 procedían de áreas no quirúrgicas (35.76%); y 30 pacientes estudiados estaban hospitalizados en áreas quirúrgicas (14/151) o UCI del hospital (22/151, 14.5%).
Todas las muestras fueron procesadas según el algoritmo diagnóstico para el estudio de bacteriemia de nuestro laboratorio: tinción de gran a los frascos positivos y posterior reaislamiento en los medios de cultivo adecuados. La identificación de los microorganismos se realizó mediante WIDER® para bacilos GRAM negativo y cocos GRAM positivos (F. Soria-Melguizo), VITEK-2® para levaduras (Biomérieaux); y API ANA para bacterias anaerobias (Biomérieaux).Para la identificación de estreptococos y neumococos se utilizó la prueba de susceptibilidad a la bacitracina y a la optoquina.
De forma paralela, los frascos positivos se utilizaron para su procesamiento directo para identificación por el sistema de espectrometría de masas MALDITOF-MS® -MS (Bruker), utilizando el kit Sepsityper® (Bruker), para ello utilizamos 1 ml de hemocultivo, lo sometemos a un proceso de lisis con solución LYSIS BUFFER (LB), centrifugamos y lavamos con la solución WASHING BUFFER (WB). Posteriormente se añade al pellet 300 µl de agua HPLC y 900 µl de alcohol. Centrifugamos el eppendorf a 16000 rpm y añadimos ácido fórmico y acetonitrilo. Finalmente centrifugar y depositar 1µl del sobrenadante en la pletina proporcionada por el fabricante. Añadir 1µl de matriz (ácido -alfa-ciano-4-hidroxicinámico) y hacer la lectura automatizada en el sistema MS MALDITOF® Autoflex III (Bruker Daltonics), el cual posee un software (FlexAnalysis Biotyper 3.3 (Bruker Daltonik) que analiza los distintos perfiles proteínicos obtenidos y puntúa automáticamente con scores de 0 a 3 en función de la fiabilidad de la identificación obtenida, siendo considerada como una identificación no fiable a nivel de género ni de especie del microorganismo cuando se obtiene un score inferior al 1,699. Identificación probable a nivel de género si se obtiene un score entre 1,700 y 1,999. Identificación segura a nivel de género y probable a nivel de especie si el score está comprendido entre 2,000 y 2,299. Y alta probabilidad de identificación de especie si el score está comprendido entre 2,300 y 3,000.
Los datos obtenidos se han analizado mediante distribución de frecuencias absolutas y relativas. Para las variables continuas se presenta el número de observaciones válidas así como estadísticas que describan el promedio y la distribución de la distribución (media ± desviación estándar). Para la estimación de la correlación entre técnicas se estima el Índice de Correlación Kappa de los resultados obtenidos por ambas técnicas, el cual establece una fortaleza de concordancia de resultados excelente cuando el índice es de 0.81-1, bueno si es entre 0.61-0.8, moderado si 0.41-0.6, ligero si 0.21-0.4, y malo si menor de 0.2.
Resultados
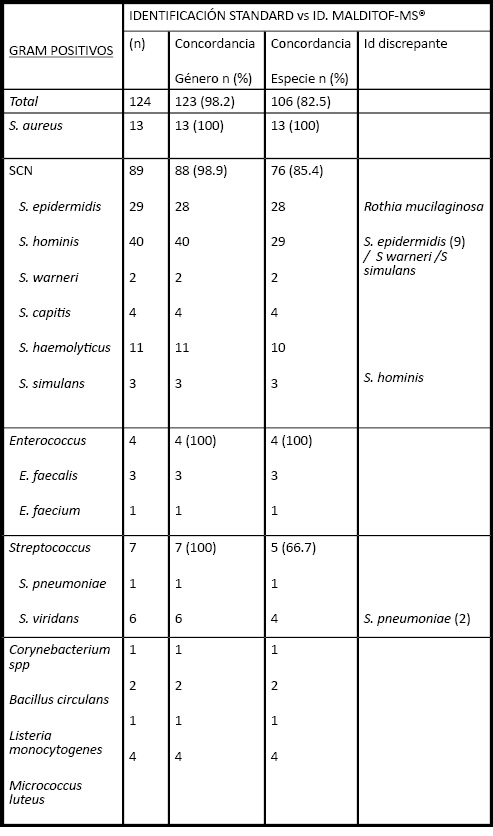
La distribución de los aislamientos estudiados en los 166 pacientes fue de un 25.3% de Bacilos GRAM Negativos (BGN, n=42), y de un 74.7% de Cocos GRAM Positivos (CGP, n=124) de los cuales 89 fueron bacteriemias causadas por Estafilococos coagulasa negativo (ECN), 13 por S. aureus, 4 por microorganismos pertenecientes al género Enterococcus, 6 pertenecieron al grupo St. viridans y 1 St. pneumoniae. Finalmente se diagnosticaron 11 bacteriemias causadas por otros microorganismos GRAM positivo: 1 Corynebacterium spp, 2 casos de Bacillus circulans, 1 bacteriemia por Listeria monocytogenes; 4 por Micrococcus luteus; 2 Clostridium perfringens y un Propinebacterium acnés.
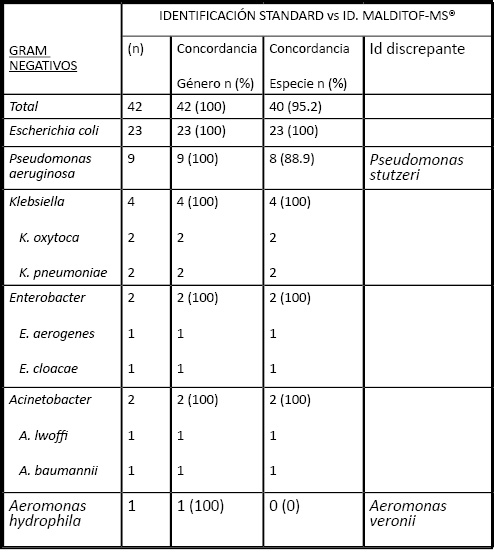
La concordancia global de resultados entre la identificación por MS MALDITOF® y la identificación tradicional ha sido del 98.7% respecto al género de los microorganismos implicados, y del 95.8 % respecto a su especie. Estos datos, cuando se separan entre microorganismos GRAM negativos (GN) y GRAM positivos (GP), pasan a 100% de concordancia en género en los GN (95.2% de concordancia en especie); y del 98.2% de concordancia en género en GP (82.5% en especie).
La correlación en la identificación de los bacilos GRAM negativo fue excelente, tanto para el género como para la especie. Sólo hubo un caso en el que la ID convencional (Wider) fue Aeromonas hydrophila mientras que la identificación por MALDITOF-MS® fue Aeromonas veronii (14).
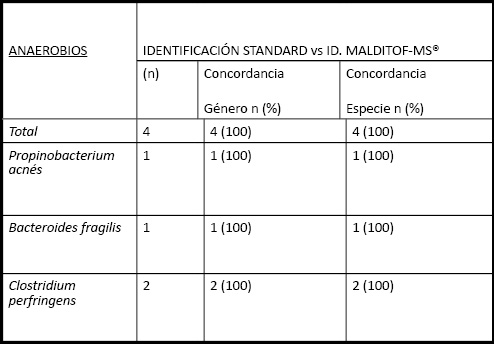
Respecto a la identificación de los microorganismos GRAM positivos, destaca la concordancia del 100% en todos los casos de Staphylococcus aureus (13/13) (15), y Enterococcus (4/4) (16). Sin embargo, dentro del grupo de Estafilococos Coagulasa Negativo (ECN) (17), el porcentaje de correlación de identificación en género fue del 98.9% debido a una discrepancia, donde el sistema MALDITOF-MS® identificó como Rothia mucilaginosa, mientras que la identificación convencional a través de identificación automatizada por sistema WIDER® lo identificó como S epidermidis. Respecto a la identificación a nivel de especie en ECN, la concordancia fue del 86.5% debido a que el sistema WIDER® identificó 11 casos de bacteriemia por Staphylococcus hominis, mientras que MALDITOF-MS® identificó 9 casos de Staphylococcus epidermidis, 1 caso de Staphylococcus warnerii, y otro caso de Staphylococcus simulans. Finalmente MALDITOF-MS® diagnóstico un caso S hominis que por el contrario el sistema WIDER® lo identificó como S haemolyticus. En relación al género Streptococcus, la identificación a nivel de especie fue del 66.7%, debido a que de los dos casos en los que la identificación por MALDITOF-MS® resultó ser Streptococcus pneumoniae, dicha identificación no se correlacionó con la identificación realizada mediante métodos bioquímicos tales como la sensibilidad en disco-placa a la optoquina y la bacitracina, las cuales fueron complementadas con baterías comerciales API-Strept® (Biomérieaux). Respecto a los aislamientos de bacilos GRAM positivo (BGP), solo analizamos 4 casos de BGP aerobios (1 Corynebacterium spp, 2 casos de Bacillus circulans, 1 bacteriemia por Listeria monocytogenes); y 3 casos de BGP anaeróbicos (2 Clostridium perfringens y un Propinebacterium acnés). En todos los casos la concordancia de resultados tanto en género como en especie fue del 100% por ambos sistemas de identificación. Todos los datos del estudio de concordancia se describen en la Tabla1.
Tabla 1. Porcentajes de concordancia de resultados entre los dos sistemas de identificación automatizada en evaluación: Sistema de identificación automatizada WIDER / VITEK2 vs Espectrometría de masas MALDITOF MS®.
Nuestra principal limitación a la hora de obtener un resultado tras análisis por espectrometría de masas MALDI-TOF MS ha sido la no obtención del pellet tras la centrifugación del mililitro de hemocultivo positivo junto a la solución Lysis buffer. En nuestro estudio esto sólo ha ocurrido en 5 casos (1,3%), no obteniendo finalmente una identificación del microorganismo por medio de MALDI-TOF MS.
Discusión
La importancia de la mortalidad asociada al retraso en el inicio de un tratamiento antibiótico correcto en la actualidad condiciona al microbiólogo clínico a adaptar sus protocolos diagnósticos, en especial en el caso de la bacteriemia. La rapidez y relativa sencillez del uso de la espectrometría de masas ya ha demostrado su utilidad en comparación con otros sistemas diagnósticos en el laboratorio de microbiología (18-21). El diagnóstico del agente etiológico productor de la bacteriemia requiere el cultivo previo del microorganismo en medio sólido desde los frascos de hemocultivo positivo, lo que supone un retraso de al menos 8-24 horas. Aunque la tinción de gram del hemocultivo positivo es informativa, la posibilidad de disponer en ese mismo momento de la identificación de género y especie supone un gran avance, que tiene una inestimable aplicación tanto en la clínica como en la gestión de los costes en antibioterapia y en días de ingreso hospitalario (22). Con este método de trabajo hemos conseguido adelantar el resultado de la identificación del microorganismo causante de bacteriemia en 17 a 23 horas durante la jornada laboral y de 13 a 16 horas en periodo de continuidad asistencial. Siendo nuestro volumen de trabajo de una media de 7 frascos de hemocultivo positivos al día durante la jornada laboral y 2 durante el periodo de continuidad asistencial, utilizamos este nuevo método de identificación en un frasco de hemocultivo positivo (aeróbico y anaeróbico) de una de las distintas tomas de cada paciente (una, dos o tres tomas de hemocultivo). Reaislando el resto de frascos de las distintas tomas en medios de cultivo para su posterior identificación mediante pruebas bioquímicas convencionales.
En nuestro estudio hemos implementado un nuevo método para el tratamiento de los hemocultivos positivos, como paso previo a su análisis mediante MALDITOF-MS®. Hasta el momento, diversos autores han mostrado los resultados de la aplicación de diferentes protocolos para procesar el hemocultivo positivo (23), que incluyen desde el volumen de sangre de hemocultivo utilizado y la centrifugación hasta diversos protocolos de extracción, sin un notable grado de estandarización.
Los resultados de nuestro trabajo muestran un grado de concordancia excelente respecto otros métodos de identificación, destacando su utilidad en todos los casos de bacteriemias causadas por bacilos GRAM-negativos, Staphylococcus aureus, microorganismos del género Enterococcus, y bacterias anaeróbicas. No obstante hay que remarcar las limitaciones de esta tecnología en dos situaciones clínicas: la primera, de gran trascendencia, la bacteriemia por Streptococcus pneumoniae y la posibilidad de confusión por parte del sistema MALDITOF MS® con Streptococcus del grupo viridans (18,24). Este fallo, bien conocido del sistema MALDITOF-MS®, se debe según algunos autores, a la gran similitud a nivel proteómico de múltiples especies de streptococcus, hasta el punto de ser necesario hacer genotipado molecular de las mismas para su correcta diferenciación (25); la segunda, de mucha menor trascendencia clínica, es el caso de la asignación de especie para los estafilococos coagulasa negativos, en los que el porcentaje de concordancia en especie desciende al 86.5%. Hay que destacar, que en ningún caso hubo discordancias con la identificación de Staphylococcus aureus, y en todos los casos de discrepancia a nivel de especie no supuso ningún cambio en el manejo clínico del paciente. Queda aún por esclarecer si la identificación correcta en estos casos es atribuible a errores en la tecnología MALDITOF-MS® o por el contrario se deben a errores en la identificación convencional. La secuenciación del ARN16S y el posterior análisis filogenético de las secuencias habría permitido establecer cuál de los dos sistemas estaba emitiendo el resultado correcto (25).
En resumen, presentamos un estudio en el que se utiliza el kit Sepsityper (Bruker) y la espectrometría de masas MALDITOF-MS® para la identificación de patógenos directamente a partir de frascos de hemocultivos positivos. Esta es una herramienta rápida y fiable para la identificación del agente etiológico responsable de bacteriemia en menos de 30 minutos, y sus hallazgos aportan una importante información para la toma de decisiones acerca de la antibioterapia empírica instaurada.
REFERENCIAS
- Kollef MH, Sherman G, Ward S, Fraser VJ. Inadequate antimicrobial treatment of infections. A risk factor for hospital mortality among critically ill patients. Chest. 1999; 115: 462-74.
- Kollef M. Appropriate empirical antibacterial therapy for nosocomial infections: getting it right the first time. Drugs. 2003; 63:2157-68.
- Leibovici L, et al. The benefit of appropriate empirical antibiotic treatment in patients with bloodstream infection. J Intern Med 1998; 244: 379-86.
- Ibrahim EH, Sherman G, Ward S, Fraser V, Kollef M. The influence of inadequate antimicrobial treatment of bloodstream infections on patient outcomes in the ICU setting. Chest. 2000; 118: 146-55.
- Anhalt JP, Fenselau C. Identification of bacteria using mass spectrometry. Anal Chem. 1975; 47:219-25.
- Holland RD, Wilkes JG, Rafii F, Sutherland JB, Persons CC, Voorhees KJ, et-al. Rapid identification of intact whole bacteria based on spectral patterns using matrix-assisted laser desorption/ionization with time-of-flight mass spectrometry. Rapid Commun Mass Spectrom. 1996; 10:1227-32.
- Krishnamurthy T, Ross P.L. Rapid identification of bacteria by direct matrix-assisted laser desorption/ionization mass spectrometry analysis of whole cells. Rapid Commun Mass Spectrom. 1996; 10:1992-6.
- Fenselau C, Demirev P.A. Characterization of intact microorganisms by MALDI mass spectrometry. Mass Spectrom Rev. 2001;20:157-71.
- Seng P, Drancourt M, Gouriet F, La Scola B, Fournier PE, Rolain JM, et-al. Ongoing revolution in bacteriology: Routine identification of bacteria by matrix-assisted laser desorption ionization time-of-flight mass spectrometry. Clin Infect Dis. 2009; 49:543-51.
- E Bessède, M. Angla-gre, Y. Delagarde, S. Sep Hieng, A. Ménard and F. Mégraud. Matrix-assisted laser-desorption/ionization BIOTYPER: experience in the routine of a University hospital. Clin Microbiol Infect 2011; 17: 533–538.
- Seng P, Drancourt M, Gouriet F et al. Ongoing revolution in bacteriology: routine identification of bacteria by matrix-assisted laser desorption ionization time-of-flight mass spectrometry. Clin Infect Dis 2009; 49: 543–551.
- Stevenson LG, Drake SK, Murray PR. Rapid Identification of Bacteria in Positive Blood Culture Broths by Matrix-Assisted Laser Desorption Ionization–Time of Flight Mass Spectrometry. J Clin Microbiol. 2010 ;48:444-7.
- Martínez-Lamas L(1), Pérez del Molino ML, Pardo F, Varela E, Regueiro BJ. Espectrometria de masas matrix-assisted laser desorption ionization time-of-flight vs. metodologia convencional en la identificacion de Candida no-albicans. Enferm Infecc Microbiol Clin. 2011 Oct;29(8):568-72..
- Dallagassa CB, Surek M, Stets MI, Huergo L, Souza EM, Pedrosa FO, et al. Identification of aeromonas species by matrix-assisted laser desorption ionization-time of flight mass spectrometry (maldi-tof ms). Clinical Chemistry and Laboratory Medicine. 2011;49:S833-S.
- Szabados F, Woloszyn J, Richter C, Kaase M, Gatermann S. Identification of molecularly defined Staphylococcus aureus strains using matrix-assisted laser desorption/ionization time of flight mass spectrometry and the Biotyper 2.0 database. Journal of Medical Microbiology. 2010;59(7):787-90.
- Hinse D, Vollmer T, Erhard M, Welker M, Moore ER, Kleesiek K, et al. Differentiation of species of the Streptococcus bovis/equinus-complex by MALDI-TOF Mass Spectrometry in comparison to sodA sequence analyses. Syst Appl Microbiol. 2011 Feb;34(1):52-7.
- Dubois D, Leyssene D, Chacornac JP, Kostrzewa M, Schmit PO, Talon R, et al. Identification of a Variety of Staphylococcus Species by Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry. Journal of Clinical Microbiology. 2010;48(3):941-5.
- Risch M, Radjenovic D, Han JN, Wydler M, Nydegger U, Risch L. Microbial identification using MALDI TOF. Swiss Medical Weekly. 2010;140: w13095
- Ferreira L, Sanchez-Juanes F, Garcia-Fraile P, Rivas R, Mateos PF, Martinez-Molina E, et al. MALDI-TOF Mass Spectrometry Is a Fast and Reliable Platform for Identification and Ecological Studies of Species from Family Rhizobiaceae. Plos One. 2011;6(5).
- Cherkaoui A, Emonet S, Fernandez J, Schorderet D, Schrenzel J. Evaluation of matrix-assisted laser desorption ionization-time of flight mass spectrometry for rapid identification of Beta-hemolytic streptococci. J Clin Microbiol. 2011;49(8):3004-5.
- Seng P, Rolain JM, Fournier PE, La Scola B, Drancourt M, Raoult D. MALDI-TOF-mass spectrometry applications in clinical microbiology. Future Microbiol. 2010;5:1733-54.
- Ferreira L, Sanchez-Juanes F, Porras-Guerra I, Garcia-Garcia MI, Garcia-Sanchez JE, Gonzalez-Buitrago JM, et al. Microorganisms direct identification from blood culture by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Clin Microbiol Infect. 2011;17(4):546-51.
- Ferreira L, Sanchez-Juanes F, Munoz-Bellido JL, Gonzalez-Buitrago JM. Rapid method for direct identification of bacteria in urine and blood culture samples by matrix-assisted laser desorption ionization time-of-flight mass spectrometry: intact cell vs. extraction method. Clin Microbiol Infect. 2011 ;17(7):1007-12.
- Stevenson LG, Drake SK, Murray PR. Rapid identification of bacteria in positive blood culture broths by matrix-assisted laser desorption ionization-time of flight mass spectrometry. J Clin Microbiol. 2010 ;48(2):444-7.
- Bizzini A, Jaton K, Romo D, Bille J, Prod’hom G, Greub G. Matrix-assisted laser desorption ionization-time of flight mass spectrometry as an alternative to 16S rRNA gene sequencing for identification of difficult-to-identify bacterial strains. J Clin Microbiol. 2011;49(2):693-6.
INFORMACIÓN DEL ARTÍCULO
Autor para la correspondencia: Raquel Camacho Luque. Servicio de Microbiología. Hospital Universitario San Cecilio. Granada. Avda. Doctor Oloriz, 16. 18012 Granada, España email: raquelcluque@hotmail.com