
72
Raquel Camacho Luque
Sepsityper® para la identificación rápida de microorganismos
gativo fue excelente, tanto para el género como para la especie.
Sólo hubo un caso en el que la ID convencional (Wider) fue
Aero-
monas hydrophila
mientras que la identificación por MALDITOF-
MS® fue
Aeromonas veronii
(14).
Respecto a la identificación de los microorganismos GRAM
positivos, destaca la concordancia del 100% en todos los casos
de
S
taphylococcus
aureus (13/13)
(15), y
Enterococcus
(4/4) (16).
Sin embargo, dentro del grupo de Estafilococos Coagulasa Ne-
gativo (ECN) (17), el porcentaje de correlación de identificación
en género fue del 98.9% debido a una discrepancia, donde el
sistema MALDITOF-MS® identificó como
Rothia mucilaginosa
,
mientras que la identificación convencional a través de identi-
ficación automatizada por sistema WIDER® lo identificó como
S
epidermidis.
Respecto a la identificación a nivel de especie en
ECN, la concordancia fue del 86.5% debido a que el sistema
WIDER® identificó 11 casos de bacteriemia por
S
taphylococcus
hominis,
mientras que MALDITOF-MS® identificó 9 casos de
S
ta-
phylococcus
epidermidis,
1 caso de
S
taphylococcus
warnerii,
y
otro caso de
S
taphylococcus
simulans.
Finalmente MALDITOF-
MS® diagnóstico un caso
S hominis que
por el contrario
el sis-
tema WIDER® lo identificó como
S haemolyticus.
En relación al
género
Streptococcus
, la identificación a nivel de especie fue del
66.7%, debido a que de los dos casos en los que la identificación
por MALDITOF-MS® resultó ser
Streptococcus pneumoniae
, di-
cha identificación no se correlacionó con la identificación reali-
zada mediante métodos bioquímicos tales como la sensibilidad
en disco-placa a la optoquina y la bacitracina, las cuales fueron
complementadas con baterías comerciales API-Strept® (Biomé-
rieaux). Respecto a los aislamientos de bacilos GRAM positivo
(BGP), solo analizamos 4 casos de BGP aerobios (1
Corynebacte-
rium spp
, 2 casos de
Bacillus circulans
, 1 bacteriemia por
Listeria
monocytogenes
); y 3 casos de BGP anaeróbicos (2
Clostridium
perfringens
y un
Propinebacterium acnés)
. En todos los casos la
concordancia de resultados tanto en género como en especie fue
del 100% por ambos sistemas de identificación. Todos los datos
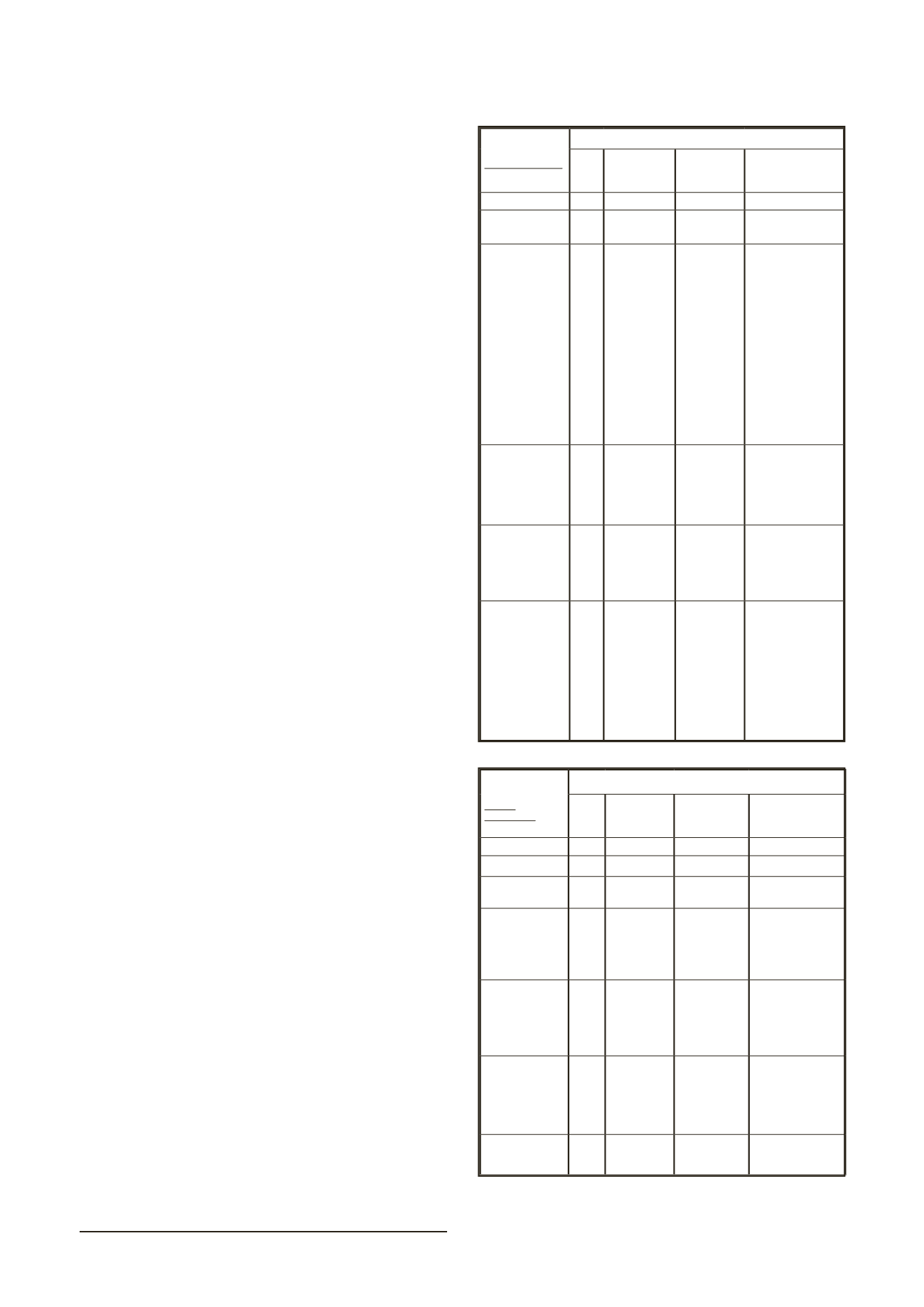
del estudio de concordancia se describen en la Tabla1.
Nuestra principal limitación a la hora de obtener un resulta-
do tras análisis por espectrometría de masas MALDI-TOF MS ha
sido la no obtención del pellet tras la centrifugación del mililitro
de hemocultivo positivo junto a la solución Lysis buffer. En nues-
tro estudio esto sólo ha ocurrido en 5 casos (1,3%), no obtenien-
do finalmente una identificación del microorganismo por medio
de MALDI-TOF MS.
DISCUSIÓN
La importancia de la mortalidad asociada al retraso en el
inicio de un tratamiento antibiótico correcto en la actualidad
condiciona al microbiólogo clínico a adaptar sus protocolos diag-
nósticos, en especial en el caso de la bacteriemia. La rapidez y
relativa sencillez del uso de la espectrometría de masas ya ha
demostrado su utilidad en comparación con otros sistemas diag-
nósticos en el laboratorio de microbiología (18-21). El diagnós-
tico del agente etiológico productor de la bacteriemia requiere
el cultivo previo del microorganismo en medio sólido desde los
frascos de hemocultivo positivo, lo que supone un retraso de al
menos 8-24 horas. Aunque la tinción de gram del hemocultivo
positivo es informativa, la posibilidad de disponer en ese mis-
mo momento de la identificación de género y especie supone
un gran avance, que tiene una inestimable aplicación tanto en
la clínica como en la gestión de los costes en antibioterapia y
en días de ingreso hospitalario (22). Con este método de traba-
jo hemos conseguido adelantar el resultado de la identificación
del microorganismo causante de bacteriemia en 17 a 23 horas
durante la jornada laboral y de 13 a 16 horas en periodo de con-
tinuidad asistencial. Siendo nuestro volumen de trabajo de una
media de 7 frascos de hemocultivo positivos al día durante la
jornada laboral y 2 durante el periodo de continuidad asisten-
cial, utilizamos este nuevo método de identificación en un frasco
de hemocultivo positivo (aeróbico y anaeróbico) de una de las
distintas tomas de cada paciente (una, dos o tres tomas de he-
GRAM POSITIVOS
IDENTIFICACIÓN STANDARD vs ID. MALDITOF-MS®
(n)
Concordancia
Género n (%)
Concordancia
Especie n (%)
Id discrepante
Total
124 123 (98.2)
106 (82.5)
S. aureus
13 13 (100)
13 (100)
SCN
S. epidermidis
S. hominis
S. warneri
S. capitis
S. haemolyticus
S. simulans
89
29
40
2
4
11
3
88 (98.9)
28
40
2
4
11
3
76 (85.4)
28
29
2
4
10
3
Rothia mucilaginosa
S. epidermidis
(9)
/ S warneri /S
simulans
S. hominis
Enterococcus
E. faecalis
E. faecium
4
3
1
4 (100)
3
1
4 (100)
3
1
Streptococcus
S. pneumoniae
S. viridans
7
1
6
7 (100)
1
6
5 (66.7)
1
4
S. pneumoniae
(2)
Corynebacterium
spp
Bacillus circulans
Listeria
monocytogenes
Micrococcus
luteus
1
2
1
4
1
2
1
4
1
2
1
4
GRAM
NEGATIVOS
IDENTIFICACIÓN STANDARD vs ID. MALDITOF-MS®
(n)
Concordancia
Género n (%)
Concordancia
Especie n (%)
Id discrepante
Total
42 42 (100)
40 (95.2)
Escherichia coli
23 23 (100)
23 (100)
Pseudomonas
aeruginosa
9
9 (100)
8 (88.9)
Pseudomonas
stutzeri
Klebsiella
K. oxytoca
K. pneumoniae
4
2
2
4 (100)
2
2
4 (100)
2
2
Enterobacter
E. aerogenes
E. cloacae
2
1
1
2 (100)
1
1
2 (100)
1
1
Acinetobacter
A. lwoffi
A. baumannii
2
1
1
2 (100)
1
1
2 (100)
1
1
Aeromonas
hydrophila
1 1 (100)
0 (0)
Aeromonas
veronii

















