
73
Raquel Camacho Luque
Sepsityper® para la identificación rápida de microorganismos
mocultivo). Reaislando el resto de frascos de las distintas tomas
en medios de cultivo para su posterior identificación mediante
pruebas bioquímicas convencionales.
En nuestro estudio hemos implementado un nuevo mé-
todo para el tratamiento de los hemocultivos positivos, como
paso previo a su análisis mediante MALDITOF-MS®. Hasta el
momento, diversos autores han mostrado los resultados de la
aplicación de diferentes protocolos para procesar el hemocul-
tivo positivo (23), que incluyen desde el volumen de sangre de
hemocultivo utilizado y la centrifugación hasta diversos proto-
colos de extracción, sin un notable grado de estandarización.
Los resultados de nuestro trabajo muestran un grado de
concordancia excelente respecto otros métodos de identifica-
ción, destacando su utilidad en todos los casos de bacteriemias
causadas por bacilos GRAM-negativos, Staphylococcus aureus,
microorganismos del género Enterococcus, y bacterias anaeró-
bicas. No obstante hay que remarcar las limitaciones de esta
tecnología en dos situaciones clínicas: la primera, de gran tras-
cendencia, la bacteriemia por
Streptococcus pneumoniae
y la
posibilidad de confusión por parte del sistema MALDITOF MS®
con
Streptococcus del grupo viridans
(18,24). Este fallo, bien
conocido del sistema MALDITOF-MS®, se debe según algunos
autores, a la gran similitud a nivel proteómico de múltiples
especies de streptococcus, hasta el punto de ser necesario
hacer genotipado molecular de las mismas para su correcta
diferenciación (25); la segunda, de mucha menor trascenden-
cia clínica, es el caso de la asignación de especie para los es-
tafilococos coagulasa negativos, en los que el porcentaje de
concordancia en especie desciende al 86.5%. Hay que destacar,
que en ningún caso hubo discordancias con la identificación de
Sta
phylococcus aureus
, y en todos los casos de discrepancia a
nivel de especie no supuso ningún cambio en el manejo clíni-
co del paciente. Queda aún por esclarecer si la identificación
correcta en estos casos es atribuible a errores en la tecnolo-
gía MALDITOF-MS® o por el contrario se deben a errores en la
identificación convencional. La secuenciación del ARN16S y el
posterior análisis filogenético de las secuencias habría permi-
tido establecer cuál de los dos sistemas estaba emitiendo el
resultado correcto (25).
En resumen, presentamos un estudio en el que se utiliza el
kit Sepsityper (Bruker) y la espectrometría de masas MALDITOF-
MS® para la identificación de patógenos directamente a partir
de frascos de hemocultivos positivos. Esta es una herramienta
rápida y fiable para la identificación del agente etiológico res-
ponsable de bacteriemia en menos de 30 minutos, y sus hallaz-
gos aportan una importante información para la toma de deci-
siones acerca de la antibioterapia empírica instaurada.
REFERENCIAS
1.
Kollef MH, Sherman G, Ward S, Fraser VJ. Inadequate
antimicrobial treatment of infections. A risk factor for hospital
mortality among critically ill patients. Chest. 1999; 115: 462-74.
2.
Kollef M. Appropriate empirical antibacterial therapy for
nosocomial infections: getting it right the first time. Drugs.
2003; 63:2157-68.
3.
Leibovici L, et al. The benefit of appropriate empirical antibiotic
treatment in patients with bloodstream infection. J Intern Med
1998; 244: 379-86.
4.
IbrahimEH, ShermanG,Ward S, Fraser V, KollefM. The influence
of inadequate antimicrobial treatment of bloodstream
infections on patient outcomes in the ICU setting. Chest. 2000;
118: 146-55.
5.
Anhalt JP, Fenselau C. Identification of bacteria using mass
spectrometry. Anal Chem. 1975; 47:219-25.
6.
Holland RD, Wilkes JG, Rafii F, Sutherland JB, Persons CC,
Voorhees KJ, et-al. Rapid identification of intact whole
bacteria based on spectral patterns using matrix-assisted laser
desorption/ionization with time-of-flight mass spectrometry.
Rapid Commun Mass Spectrom. 1996; 10:1227-32.
7.
Krishnamurthy T, Ross P.L. Rapid identification of bacteria
by direct matrix-assisted laser desorption/ionization mass
spectrometry analysis of whole cells. Rapid Commun Mass
Spectrom. 1996; 10:1992-6.
8.
Fenselau C, Demirev P.A. Characterization of intact
microorganisms by MALDI mass spectrometry. Mass Spectrom
Rev. 2001;20:157-71.
9.
Seng P, Drancourt M, Gouriet F, La Scola B, Fournier PE,
Rolain JM, et-al. Ongoing revolution in bacteriology: Routine
identification of bacteria by matrix-assisted laser desorption
ionization time-of-flight mass spectrometry. Clin Infect Dis.
2009; 49:543-51.
10. E Bessède, M. Angla-gre, Y. Delagarde, S. Sep Hieng, A. Ménard
and F. Mégraud. Matrix-assisted laser-desorption/ionization
BIOTYPER: experience in the routine of a University hospital.
Clin Microbiol Infect 2011; 17: 533–538.
11. Seng P, Drancourt M, Gouriet F et al. Ongoing revolution in
bacteriology: routine identification of bacteria by matrix-
assisted laser desorption ionization time-of-flight mass
spectrometry. Clin Infect Dis 2009; 49: 543–551.
12. Stevenson LG, Drake SK, Murray PR. Rapid Identification of
Bacteria in Positive Blood Culture Broths by Matrix-Assisted
Laser Desorption Ionization–Time of Flight Mass Spectrometry.
J Clin Microbiol. 2010 ;48:444-7.
13. Martínez-Lamas L(1), Pérez del Molino ML, Pardo F, Varela
E, Regueiro BJ. Espectrometria de masas matrix-assisted
laser desorption ionization time-of-flight vs. metodologia
convencional en la identificacion de Candida no-albicans.
Enferm Infecc Microbiol Clin. 2011 Oct;29(8):568-72..
14. Dallagassa CB, Surek M, Stets MI, Huergo L, Souza EM, Pedrosa
FO, et al. Identification of aeromonas species by matrix-assisted
laser desorption ionization-time of flight mass spectrometry
(maldi-tof ms). Clinical Chemistry and Laboratory Medicine.
2011;49:S833-S.
15. Szabados F, Woloszyn J, Richter C, Kaase M, Gatermann S.
Identification of molecularly defined Staphylococcus aureus
strains using matrix-assisted laser desorption/ionization time
of flight mass spectrometry and the Biotyper 2.0 database.
Journal of Medical Microbiology. 2010;59(7):787-90.
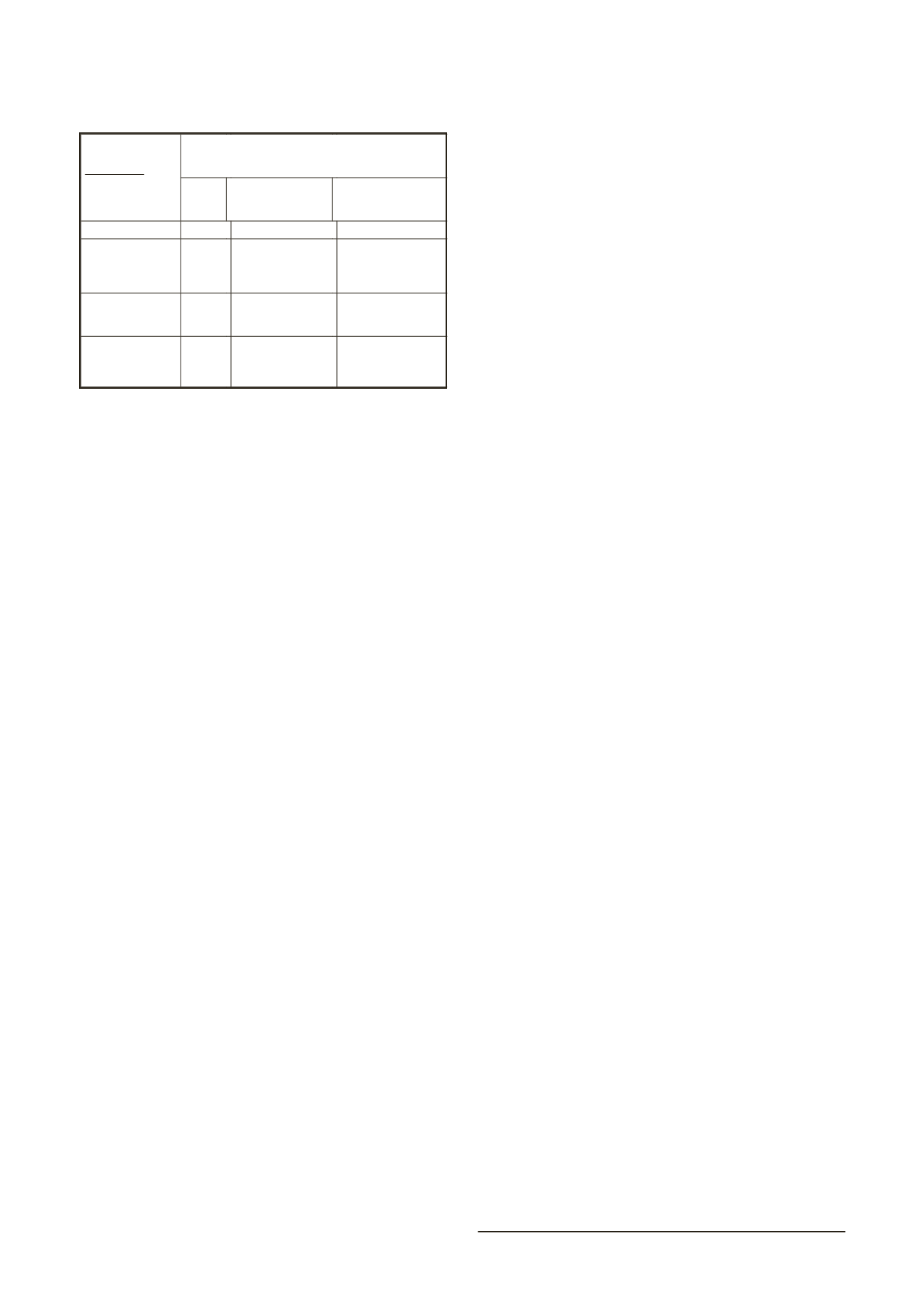
Tabla 1. Porcentajes de concordancia de resultados entre los dos
sistemas de identificación automatizada en evaluación: Sistema
de identificación automatizada WIDER / VITEK2 vs Espectrometría
de masas MALDITOF MS®.
ANAEROBIOS
IDENTIFICACIÓN STANDARD vs ID. MALDITOF-MS®
(n)
Concordancia
Género n (%)
Concordancia
Especie n (%)
Total
4
4 (100)
4 (100)
Propinobacterium
acnés
1
1 (100)
1 (100)
Bacteroides fragilis
1
1 (100)
1 (100)
Clostridium
perfringens
2
2 (100)
2 (100)

















